-
阿尔兹海默症(Alzheimer disease,AD)是一种老年性神经退化性疾病,主要临床表现为认知功能下降、记忆力逐渐丧失、精神和日常生活能力显著降低。目前全球AD患者超过千万,且预计在未来的几十年内其患病率将急剧增长[1]。
AD作为一种不可逆神经退行性疾病,发病机制十分复杂,涉及到病理、生理系统间的各种相互作用,主要的病因包括脑灰质中β-淀粉样蛋白积聚的淀粉样蛋白斑块和与tau蛋白相关的神经纤维缠结[2]。有研究表明,AD的疾病进程与其他类型的分子病理、生理机制也存在相关性,如突触功能障碍、神经炎症和脑代谢功能紊乱等[3-5]。因此,明确AD的发病进程和病理、生理学特征是一项十分艰巨的挑战。
近年来随着人工智能技术的兴起,各种数据挖掘技术被广泛地应用于AD早期诊断和对其机理的研究中,其中,基于图论的复杂网络分析方法为临床工作者提供了一个全新的视角[6]。该方法将大脑看作一个复杂的交互网络系统,将脑区或神经元组视作网络节点,通过对比观察AD患者和正常人群中脑网络参数变化,实现对AD的早期鉴别和机理研究。在过去的十几年,从动物模型到活体人脑的跨物种研究已经揭示了AD会造成脑网络结构和功能变化,如小世界特性、富人俱乐部和分层模块化等特性的变化[7-10]。而随着诸多神经影像学技术[如结构磁共振成像(structural magnetic resonance imaging,sMRI)、功能磁共振成像(functional magnetic resonance imaging, fMRI)、PET等]在AD临床的大规模普及应用,基于神经影像的AD复杂脑网络研究成为可能[7]。
综上,复杂网络分析技术与脑神经成像技术的结合为AD的早期诊断和明确其病理机制提供了全新的思路[8-9]。笔者将基于图论复杂脑网络的相关概念,对其在AD领域的应用进展和未来发展趋势等方面进行综述。
HTML
-
自Watts和Strogatz[10]首次提出图论结合神经系统分析框架后,基于图论的复杂网络分析技术被广泛地应用于动物和人类的神经系统研究。与神经元的结构相类似,复杂网络分析试图理解由个体的功能单元(通常称为节点)及其之间的相互作用或连接(通常称为边)组成的脑结构或功能系统。这些节点及其相互作用的边结合形成一个网络模型,这个网络模型可用于描述、解释或预测它所代表的真实物理网络行为。例如,可使用N×N邻接矩阵来描述成对节点之间的相互作用关系,其中N是网络中的节点的数量,邻接矩阵中第{i,j}个元素代表了节点i和节点j之间的连接强度[11-12];并使用数学方法来表征邻接矩阵的体系结构[13]。其他类似的方法,包括超图(边可以链接任意数量的顶点)、单纯复形(高阶交互项成为基本单位)和非基于图的技术,同样可用于分析构成生物脑组织的神经元网络[14-15]。
复杂脑网络分析中,由MRI技术(如sMRI和弥散张量成像等结构成像)构建的复杂网络被称为结构性脑网络,该网络可根据成像出的脑解剖信息揭示脑结构之间的生理相互关系;使用fMRI和PET等功能性成像技术构建的复杂网络被称为功能性脑网络,通过分析该网络各节点之间的脑血流动力学或脑葡萄糖代谢水平相关性等,可以反映大脑在进行功能活动中各脑区或神经元组之间的联系。
为了实现脑网络构建,首先应对脑网络节点进行定义,现有研究多使用先验解剖模板,如标准的大脑解剖标记模板等;而各节点之间的相互关系有许多度量方法,其中使用最广泛的包括皮尔逊相关和格兰杰因果相关等[8-9]。
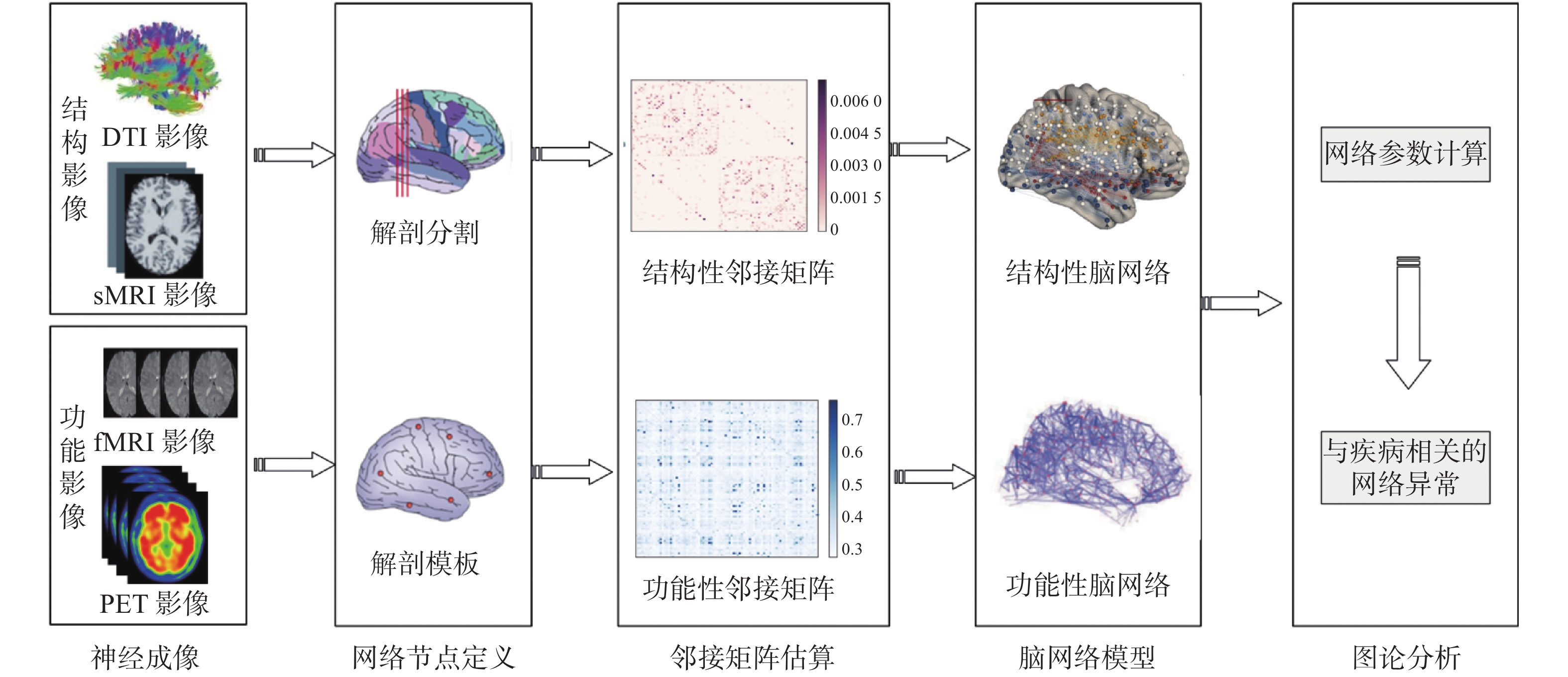
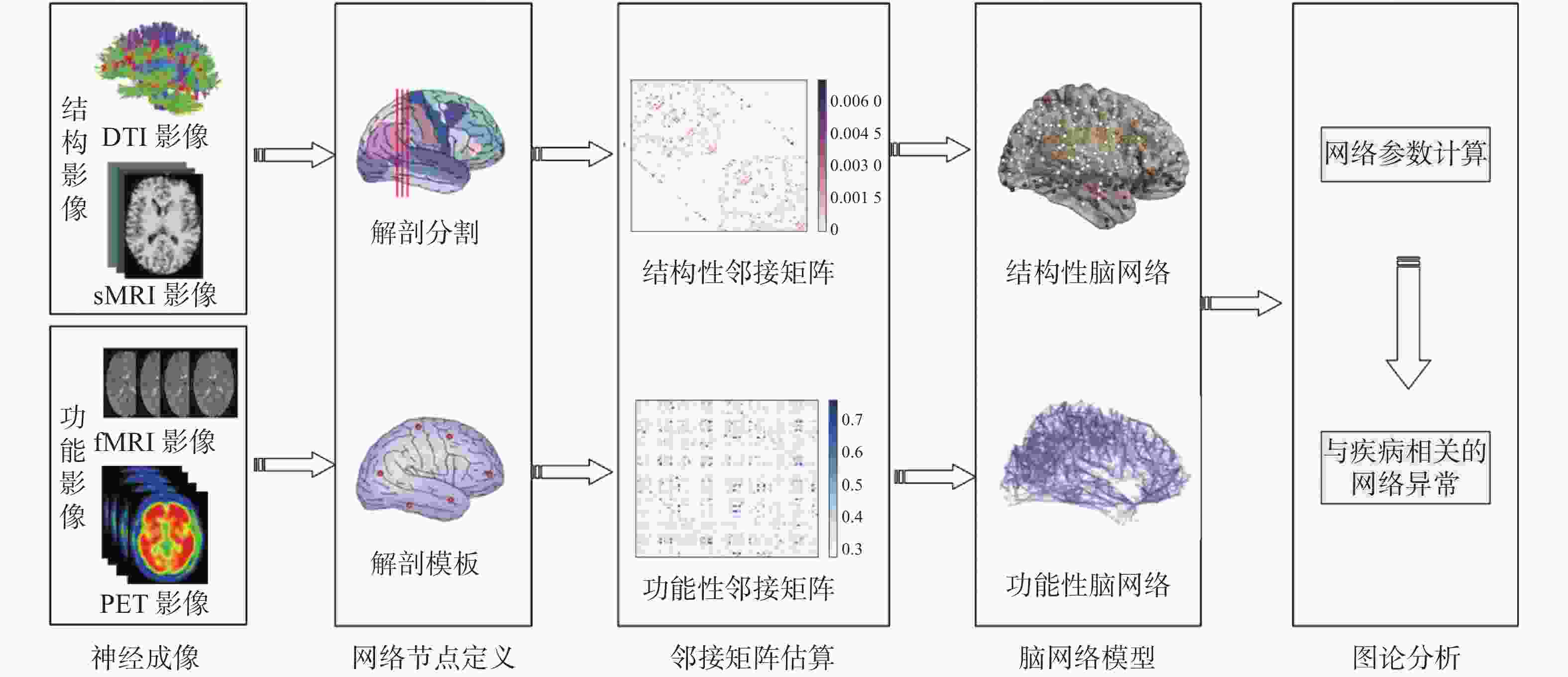
常规的复杂脑网络分析可以简要概括为以下5个步骤,如图1所示。
(1)神经成像。脑网络构建分为两种:针对结构成像如sMRI、DTI等构建结构脑网络;或针对功能成像如fMRI、PET等构建功能脑网络。
(2)脑网络节点定义。对于脑电图或脑磁图数据,可以定义为电极的位置;对于sMRI、fMRI或PET数据,可以定义为解剖学脑区。
(3)定义节点的结构或功能连接。连接可以是sMRI数据中两个脑区间之间的皮层厚度或灰质体积测量值的相关性、弥散张量成像数据中两个脑区之间的连接概率、脑电图数据中两个脑电图电极信号之间格兰杰因果关系或相位同步性、fMRI数据中两脑区之间血流动力信号的因果相关性、PET数据中两脑区之间的代谢相关性等。
(4)计算成对节点之间的连接权重得到邻接矩阵,通常对该矩阵进行阈值化处理,去除连接较弱的连接,最后生成二值化的连接矩阵。
(5)计算脑网络模型中感兴趣的网络参数,如度、聚类系数、最短路径长度、小世界系数和模块化系数等,并将它们与随机网络的等效参数进行比较,或与对照组的网络参数进行比较,进而发现与疾病相关的网络特性。
-
AD复杂脑网络研究主要基于sMRI、fMRI、PET等神经影像(表1)。He等[16]基于sMRI影像的结构性脑网络研究结果表明,AD患者较正常对照组呈现:(1)脑网络全局整合能力下降,平均路径长度更长,全局效率降低;(2)网络局部处理能力增加,如网络具有更高的聚类系数。部分研究者将这些结果解释为AD患者脑内神经元信息分离和整合平衡发生改变,导致AD患者的脑网络倾向于向规则网络(更高聚类系数,更长路径长度)方向演变,并进一步证明了这些网络参数与认知变量和记忆表现之间具有相关性[26]。
文献 成像模态 AD患者脑结构主要发现 He等[16] sMRI 聚类系数增加,平均最短路径长度增加,小世界属性减少 Supekar等[17] fMRI 聚类系数在全局水平(全脑)和局部水平(双侧海马)减少 Zhao等[18] BOLD-fMRI 聚类系数和路径长度增加 Liu等[19] fMRI 与轻度AD患者相比,重度AD患者的fMRI振幅降低,功能连接强度减少,特别是远距离连接减少 Hahn等[20]、Bernard等[21]、Dai等[22] fMRI DMN连接减少,远距离连接减少 Huang等[23] FDG PET 颞叶脑区内的功能连接减少,额叶内和顶叶与枕叶之间的功能连接减少 Titov等[24] FDG PET 额叶和顶叶区域存在较大的功能连接减少 Duan等[25] PiB PET 小世界属性增加,集聚系数和最短路径长度减少 注:表中,AD:阿尔兹海默症;sMRI:结构磁共振成像;fMRI:功能性磁共振成像;BOLD:血氧水平依赖;FDG:氟脱氧葡萄糖;PET:正电子发射断层显像术;PiB:匹斯堡复合物;DMN:默认模式网络。 Table 1. Brain network studies for AD subjects based on neuroimaging techniques
-
Supekar等[17]基于fMRI影像的功能性脑网络研究结果表明,AD患者较正常对照组呈现:(1)脑网络路径长度显著增加,即AD患者大脑加工效率明显下降,而且该网络参数与患者疾病量表显著相关[27];(2)AD患者脑网络局部连接下降[28],如默认模式网络(静息状态下相互联系、维持健康代谢活动的若干脑区组成的网络)的连接下降。此外,虽然研究发现AD患者脑功能网络局部功能连接下降,但由于大脑具有可塑性,因此AD患者脑功能网络全局功能连接仍保持稳定[29];(3) AD患者脑网络远距离连接丢失严重[28, 30]。研究结果同样表明,中心节点在AD的形成中起着关键性作用。例如,Buckner等[31]发现,AD患者在静息态fMRI网络中心节点脑区出现较高淀粉样β-淀粉样蛋白沉积,这表明AD患者的中心节点会受到疾病选择性攻击。部分研究结果同样表明,AD患者脑网络的中心节点连接减少,特别是在额叶和颞叶区域[32]。中心节点的改变在一定程度上解释了AD患者脑网络中观察到的较长路径长度和较低局部效率。总之,这些观察结果与AD患者脑网络小世界属性减弱和远距离连接的丢失是一致的[18, 33]。
-
虽然基于PET影像的AD复杂脑网络研究起步较晚,但近年来发展迅速。基于FDG PET影像的网络分析结果表明:使用稀疏逆协方差方法可以构建代谢脑网络,通过与正常组的对比发现,AD患者在颞叶脑区内的功能连接减少,且额叶内和顶叶与枕叶之间的功能连接也有减少[23-24]。Duan等[25]基于匹斯堡复合物PET影像的脑网络分析揭示,AD患者的代谢脑网络的小世界属性增强,集聚系数降低,最短路径长度明显缩短。此外,基于PET/MRI多模态影像的复杂网络分析也提高了AD疾病的早期预测能力[34]。
2.1. 基于sMRI的结构性AD复杂脑网络研究
2.2. 基于fMRI的功能性AD复杂脑网络研究
2.3. 基于PET的功能性AD复杂脑网络研究
-
基于神经影像的复杂脑网络研究正逐渐加深学界对AD病理的理解,而该技术的未来发展方向将主要集中在脑网络结构和功能系统化研究、脑网络宏观现象背后的微观机制研究、结合PET/fMRI的多模态脑网络研究以及结合机器学习和(或)深度学习方法的复杂脑网络研究等中。
第一,复杂脑网络分析的规范化和系统化。当前脑网络研究中,研究者常用平均最短距离、中心度、小世界属性等来刻画脑结构或功能的属性。随着对该领域的不断探索,近年来也相继有研究者提出使用相关系数和介数等参数,力求更加精准、全方位地描述脑网络[35]。然而,现有的网络参数能否全面刻画AD患者的脑网络特征,是否存在未知宏观参数能更好地描述AD患者脑网络特性,都值得进一步研究。此外,网络分层研究将是一个重要趋势,最近大量实验结果表明真实脑网络具有分层结构[36]。由于不同网络层次对应不同功能单元,因此如何选取特征量来刻画这种分层结构以及分层结构如何影响AD患者脑网络是当今颇受关注的一个问题[37]。
第二,探寻AD患者脑网络宏观现象背后的微观机制也备受关注。虽然现在对网络的无标度性质、小世界性质的微观机制已有一定了解,但是,对度相关、模块结构等更为复杂、宏观性质的微观生成机制与AD病理关联之间的探索还处于起步阶段。
第三,PET/fMRI相结合的AD患者脑网络研究。随着影像学技术的发展,基于PET/fMRI成像的网络分析正在成为脑网络分析的主流方法。PET/fMRI的结合研究中,有不少研究关注PET检测的局部脑活动(常用的指标是每个体素的测量值)与fMRI网络属性的关联性分析[38-39]。因此,如何充分发挥PET/fMRI的成像优势,也是AD患者脑网络研究的重要方向之一。
第四,结合机器学习和(或)深度学习方法的脑网络分析研究。在过去的研究中,基于机器学习和(或)深度学习的分类与预测模型已广泛应用到AD早期诊断,模型框架主要包括:基于机器学习和(或)深度学习的影像特征提取;基于回归模型的特征降维、选择以及疾病分类与预测。例如,该框架在轻度认知障碍的转换预测以及AD、轻度认知障碍、正常控制组的分类中获得了较高的准确率,其中支持向量机是最常见的分类与预测模型[38-40]。今后,是否能通过复杂网络分析获取高维度影像特征,并结合机器学习和(或)深度学习进行疾病分类与预测,仍需要研究者进一步探索。
-
虽然复杂脑网络分析方法已经被应用于AD早期诊断和对其的病理生理学探索,但是此方面研究仍处于初级阶段,还存在许多亟待解决的难题。例如,AD患者脑网络参数改变规律无法完全解释其大脑结构和功能变化;网络分析方法不够完善;现有结果无法归纳脑功能的动态变化和结构变化之间的相互影响等。今后的研究一方面需要完善复杂脑网络分析方法,使神经影像能为临床提供反映AD患者脑结构和功能变化的更多定量指标,为AD早期诊断提供更具敏感性和特异性更高的脑网络影像学标记物;另一方面还需要深入研究AD患者网络参数改变与认知功能变化之间的关系,进一步解释AD病理机制。总之,复杂脑网络技术为临床进一步了解大脑的结构和功能提供了新的解决方法,因而基于神经影像的AD复杂脑网络研究将会带来广阔的临床应用前景。
利益冲突 本研究由署名作者按以下贡献声明独立开展,不涉及任何利益冲突。
作者贡献声明 潘野雄负责论文的撰写;蒋皆恢负责命题的提出和论文的审阅;王敏负责资料的整理和论文的修改;左传涛负责命题的提出和论文的审阅。






 DownLoad:
DownLoad: