-
雌激素受体(estrogen receptor,ER)广义上可分为两种,一类属于细胞内核受体超家族,而另一类则是细胞膜上G蛋白偶联ER,一般来说,ER特指前种。ER在不同组织中差异性地表达,并与相应的配体和辅助因子相互作用,控制着基因的协调表达,在机体的生殖发育、心血管活动、骨质代谢、行为认知等活动中发挥着重要的作用。已知ER信号通路与众多疾病有关,包括骨质疏松、神经退行性疾病、心血管疾病、肥胖、胰岛素抵抗以及多种肿瘤等。如临床中约80%的乳腺癌是雌激素依赖性的,又被称作激素敏感型或激素受体阳性肿瘤,全身雌激素抑制是治疗这类肿瘤的方法之一[1]。
目前,已知ER有ERα、ERβ和ERγ 3种亚型,而雌激素相关受体(estrogen-related receptor,ERR)还包含ERRα、ERRβ和ERRγ等。它们之间具有很高的同源性,与其他核受体相似,都包括6个结构域,从氨基端到羧基端分别为A、B、C、D、E和F结构域。其中A和B结构域含有转录激活区1(activation function 1,AF-1),可以在配体缺乏的情况下具有一定的激活基因转录的功能,但与E结构域相比较,这种选择性激活功能是微弱的。C结构域又称DNA结合区域,可以和DNA上的雌激素反应元件(estrogen response elements,ERE)相结合,核定位信号(nuclear localization signal)也位于该区域。D结构域是一个铰链区,它连接着C和E结构域。E结构域主要包括配体结合区域(ligand binding domain,LBD),此外还包含AF-2,AF-2的构型高度依赖LBD所结合的配体,E结构域在配体的存在下可以激活基因的转录。羧基端的F结构域的功能目前尚未完全清楚,发现其长度是可变的。在不同的3种亚型中,目前了解的比较多的是ERα和ERβ,而ERγ的功能还不清楚。ERα和ERβ的表达具有一定的组织差异性,ERα主要在子宫内膜、乳腺癌细胞、卵巢干细胞和下丘脑中表达,ERβ主要在肾、脑、骨、肺、小肠上皮、前列腺和内皮细胞中表达[2-3]。
ER参与多种细胞信号转导调控,并与其他细胞生长信号发生交叉对话,其中最主要的功能是通过基因机制完成的。在没有配体雌激素存在的情况下,无活性的ER主要位于细胞质中,并与热休克蛋白结合。一旦结合到雌激素,ER将转移,与热休克蛋白分离,转入到细胞核内,形成二聚体结构,并特异性地结合到DNA上的ERR上。接下来DNA-ER复合物可以招募其他转录因子,并启动下游DNA的转录,从而改变靶基因的表达并影响和调节细胞的功能[3-4]。
当ER与相应的DNA区域结合后,可以招募多种不同蛋白,这些蛋白被称作ER的辅调节因子(co-regulators),它们可以促进或者抑制相应的DNA向mRNA的转录,这些辅调节因子按其功能可分为辅激活因子(co-activators)和辅抑制因子(co-repressors)。事实上,辅调节因子在核受体通路中广泛存在,且一种辅调节因子可以在很多种核受体信号转导途径调节中发挥作用,它们通过对DNA上组蛋白及相关转录因子的修饰(组蛋白乙酰化或去乙酰化、甲基化、ATP依赖的染色体重构等),使染色质结构发生改变,从而使得转录更易于或更难进行。此外,很多辅调节因子还具有连接其他转录因子的功能[5-6]。
对ER众多辅调节因子的氨基酸序列的分析发现,在辅调节因子与ER的LBD相互作用区域中,广泛存在着一段保守的富含亮氨酸的螺旋序列LXXLL(L指亮氨酸,X指任意氨基酸)。事实上,LXXLL模体,又称核受体结合盒(nuclear receptor-box),在其他核受体与辅调节因子的相互作用中同样扮演着重要角色,它们之间的相互作用既具有一定的共性,又具有特异性。对LXXLL模体的研究不仅有助于加深研究人员对ER信号通路的理解,对基于ER的药物设计和疾病治疗也有着重要的指导意义。本文主要对LXXLL模体在ER辅调节因子中存在的广泛性、与ER相互作用的机制以及其在药物设计领域的应用价值等方面的研究进展加以综述。
-
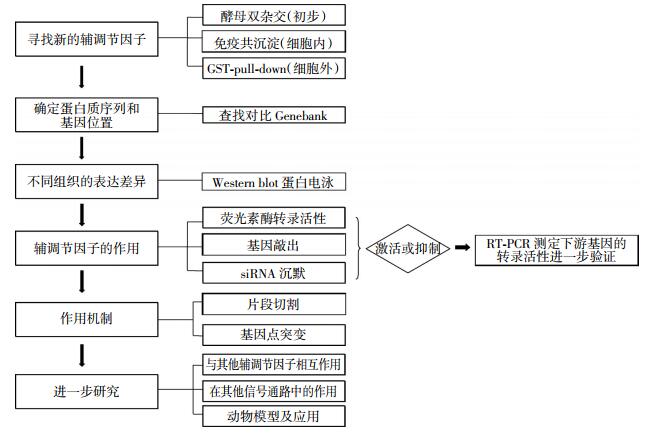
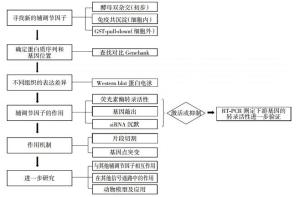
LXXLL模体最先在核受体辅激活因子CBP/p300和类固醇受体辅激活因子1(steroid receptor coactivator 1,SRC1)蛋白序列中发现,随后在甲基化CpG结合核酸内切酶1(methyl-CpG-binding endonuclease 1,MED1)、SRC/p160和过氧化物酶体增殖物激活受体γ辅激活因子1(peroxisome proliferator-activated receptor γ coactivator-1,PGC-1)等许多其他核受体辅调节因子的蛋白序列中也被广泛地发现[7]。这一研究主要是通过酵母双杂交系统(yeast two-hybrid system)、免疫共沉淀(coimmunoprecipitation)、体外报告谷胱甘肽S-转移酶沉降试验以及基因点突变(gene mutation)等技术手段完成的(图 1)。Zhang等[8]通过这类方法研究表明,位于辅调节因子SRC1和PGC-1蛋白上的LXXLL模体在调节它们与ER的相互作用中是不可或缺的。随着以上实验技术的完善和研究的深入,含有LXXLL模体的ER新的辅调节因子不断地被发现,表 1总结了目前为止已发现的含有LXXLL模体的ER辅调节因子及其功能和在相关疾病中的作用。
基因 人类基因编号 LXXLL序列(氨基酸位点) 在核受体中的作用 蛋白质功能 在疾病发生中的作用 发现时间 参考文献 CBP 1387 QLSELLRG(68-75)
QLVLLLHA(356-363)辅激活因子 组蛋白乙酰化,与SRC家族相互作用 在亨廷顿舞蹈症、白血病、脊延髓肌肉萎缩中异常表达 1996 [9] p300 2033 KLSELLRS(80-87)
QLVLLLHA(341-348)
ALQNLLRT(2050-1057)辅激活因子 组蛋白乙酰化,与SRC家族相互作用 在胃肠道肿瘤中杂合性缺失、白血病中形成异常Moz融合蛋白,突变可引起多发性肿瘤 1996 [10] SRC-1/NCOA1 8648 KLVQLLTT(252-260)
ILHRLLQE(309-318)
LLRYLLDK(368-375)
QLDELLCP(532-539)
LLQQLLTE(1054-1061)辅激活因子 组蛋白乙酰化,参与信号转导和转录激活子家族介导的转录激活 在乳腺癌、前列腺癌、子宫内膜癌中过量表达,突变可引起横纹肌肉瘤 1997 [7] SRC2/GRIP1/TIF2 10499 KLLQLLTT(640-647)
ILHRLLQD(689-696)
LLRYLLDK(744-751)
QLGRLLPN(877-884)辅激活因子 组蛋白乙酰化、与辅激活因子相关精氨酸甲基转移酶1和EP300/p300等形成复合物 在乳腺癌、脑部肿瘤中过量表达,在白血病中形成Moz-tif融合蛋白 1998 [11-13] pCAF 8850 YLFGLLRK(189-196) 辅激活因子 组蛋白乙酰化,与雌激素受体的DNA结合区域相互作用 缺失可导致直结肠癌 1998 [14-15] TRAP220/MED1/DRIP205/PIBP 5469 ILTSLLQI(603-610)
MLMNLLKD(644-651)辅激活因子 中介体,与众多MEDs形成调节复合体 缺失可导致Williams综合症 1998 [16-17] SRC3/AIB1/NCOA3 8202 KLVQLLTT(621-628)
ILHRLLQD(684-691)
LLRYLLDK(735-742)辅激活因子 组蛋白乙酰化,可与CBP/p300形成激活复合物 基因变异可导致乳腺癌、卵巢癌,在直结肠癌中过量表达 1998 [18-19] NRIF3/ITGB3BP 23421 FLKALLN(8-15)
KLDGLLEE(171-177)辅激活因子 与蛋氨酸腺苷转移酶1相互作用,调节乳腺癌细胞对雌激素的敏感程度 有待研究 1999 [20-21] ASC-1 9325 RLAVLLPG(160-167) 辅激活因子 招募组蛋白乙酰化酶,与CBP/p00、SRC-1相互作用,在核转录因子κB、激活蛋白1的转录激活中发挥重要作用 有待研究 1999 [22] LZIP/CREB3 17039 DLLALLEE(12-19)
DLLSLLSP(52-59)辅激活因子 可以与环磷酸腺苷反应元件结合,调控细胞增殖,可正向调控核转录因子κB通路,与疱疹病毒VP16感染有联系 增加白血病的迁移 2000 [23] PGC-1 10891 LLKKLLLA(143-147) 辅激活因子 参与mRNA的剪切,正向调节组蛋白乙酰化,并在能量代谢和脂肪细胞分化中具有重要作用 表达水平与乳腺癌的病死率成反比,且在心血管和糖尿病等疾病中异常表达 2000 [24-25] TRBP/RAP250/ASC-2 23054 LLVNLLQS(886-893)
LLSQLLDN(1490-1497)辅激活因子 招募组蛋白乙酰化酶,并参与DNA复制和损伤修复 突变可导致乳腺癌 2000 [26-27] DRIP150/MED14 9282 ALNILLLP(69-76)
VLTDLLPR(1181-1188)辅激活因子 中介体,与众多MEDs形成调节复合体,参与RNA聚合酶II的转录功能 有待研究 2000 [28] CIA 57727 SLINLLAD(386-393) 辅激活因子/辅抑制因子 具备氨酰基-转运RNA连接酶功能,可以与ATP结合,且与雌激素受体的结合不依赖于转录激活区2 有待研究 2001 [29] PNRC2 55629 QLKTLLKV(171-177) 辅激活因子 参与无意义密码子介导的mRNA降解 有待研究 2001 [30] TIP60 10524 MLKRLLRI(492-499) 辅激活因子 组蛋白乙酰化酶复合体NuA4的催化成分,且可以直接使毛细血管扩张共济失调突变基因乙酰化,在细胞修复中发挥作用 有待研究 2001 [31] PERC/PGC-1 133522 LLQKLLLA(155-162)
ILRELLAQ(342-349)
ILRELLAQ(342-349)辅激活因子 转录调节复合体成分之一,可以与RNA结合,参与能量代谢调节和脂肪细胞分化 基因突变可导致乳腺癌、肥胖、2型糖尿病,在甲状腺嗜酸细胞瘤中过量表达 2002 [32-33] TRAP100/MED24 9862 SLDLLLAA(447-454)
ILPGLLTD(787-794)辅激活因子 中介体,与众多MEDs形成调节复合体,参与RNA聚合酶II的转录功能 在乳腺癌中过量表达 2002 [34-35] MNAR/PELP1 27043 CLLSLLYG(28-35)
VLRDLLRY(72-79)
HLPGLLTS(94-101)
LLTSLLGL(98-105)
ELHSLLAS(181-188)
SLHTLLGA(188-195)
PLRLLLLP(281-289)
LLTHLLSD(376-383)
ELYCLLLA(496-503)
LLALLLAP(501-508)辅激活因子 可以与生长因子信号通路中组分表皮生长因子受体、肝细胞生长因子调节酪氨酸激酶作用物等相互作用,通过视网膜母细胞瘤抑制蛋白1在雌激素介导的细胞周期进展中发挥作用,并且同SRC、磷脂酰肌醇3激酶、信号转导和转录激活子3等相互作用 细胞质内浓度增高时可以导致乳腺癌,过量表达可导致子宫内膜癌、涎腺导管癌等 2004 [36-37] MLL2 8055 RLRELLIR(2685-2691)
HLDDLLNG(3037-3043)
QLQALLMQ(4221-4227)
PLOGLLGC(4252-4258)
LLQKLLRA(4187-4194)
RLRLLLTT(4714-4721)辅激活因子 具有甲基转移酶的功能,可以使组蛋白H3的Lys-3甲基化,被雌激素受体1所招募激活雌激素受体功能 缺失可导致Kabuki综合征,在乳腺癌中过量表达 2006 [38-39] CCDC62/ERAP75 84600 KLQRLLAE(633-640)
ELSTLLPI(649-656)辅激活因子 可以增强雌激素介导的cyclinD1的表达和促进细胞的生长 在前列腺癌细胞中广泛表达 2009 [40] ACTN-4 43707 KLMLLLEV(83-90) 辅激活因子 肌动蛋白结合蛋白,苯丙胺调节转录因子复合物的组成部分 突变可导致1型局灶节段性肾小球硬化 2011 [41] DAX-1 190 ILYSLLTS(145-152) 辅抑制因子 可与核受体亚家族5-A组-成员1蛋白和核受体亚家族0-B组-成员2蛋白形成多异聚体,并招募其他辅抑制子,在肾上腺激素轴的发育中具有重要作用 缺失可导致X染色体偶联的阿狄森氏病和剂量敏感性性反转 2000 [8] SHP-1 5777 ILYALLTS(19-26) 辅抑制因子 可以与DNA直接结合 突变与早发性肥胖有关 2000 [8] RIP140 8204 YLEGLLMH(20-27)
LLASLLQS(132-139)
HLKTLLKK(184-191)
QLALLLSS(265-272)
LLLHLLKS(379-386)
LLLLLLGH(499-506)
VLQLLLGN(712-719)
LLSRLLRQ(818-825)
VLKQLLLS(935-972)辅抑制因子 招募组蛋白去乙酰化酶1、组蛋白去乙酰化酶3等组蛋白去乙酰化酶,并与核受体辅激活子1形成复合物 突变可导致子宫内膜癌和不孕 2001 [42] R-MGMT 4255 VLWKLLKV(95-105) 辅抑制因子 具有甲基转移酶的活性,在DNA损伤后,可以抑制雌激素受体介导的细胞增殖 基因突变或单倍体可导致膀胱癌和脑部肿瘤,在肺癌中过量表达 2001 [43] LCoR 84458 VLSKLLMA(52-59) 辅抑制因子 可以与DNA直接结合,招募羧基末端结合蛋白1、组蛋白去乙酰化酶3、组蛋白去乙酰化酶6等共同组成转录辅抑制复合物 有待研究 2003 [44] REA 11331 ALKLLLGA(22-29) 辅抑制因子 中介体,招募组蛋白去乙酰化酶 在乳腺癌中低表达 2003 [45] ERR10/NAG7 29931 FLIWLLCW(336-343)
FLPHLLNQ(322-329)辅抑制因子 抑制雌激素受体调c-Jun氨基末端激酶琢的活性/激活蛋白,并上1/金属基质蛋白酶1信号通路 在胃肠肿瘤中高表达,可以增强鼻咽癌的侵袭能力 2004 [46] PROX1 5629 VLRKLLKR(69-76) 辅抑制因子 在机体的神经系统和内脏发育中发挥重要作用 低表达可导致肝癌、胰腺癌等 2006 [47] ERK8 9606 TLDALLPP(264-271)
LLRRLLVF(280-287)辅抑制因子 具备ATP结合活性和苏氨酸/丝氨酸激酶的活性,参与蛋白的磷酸化 有待研究 2011 [48] 注:CBP:CREB结合蛋白;SRC:类固醇受体辅激活因子;SRC-1/NCOA-1:类固醇/核受体辅激活因子1;SRC2/GRIP1:类固醇受体激活子2/糖皮质激素受体作用蛋白1;PCAF:P300/CBP相关因子;TRAP220:甲状腺激素受体相关蛋白220;MED:甲基化CpG结合核酸内切酶;DRIP205:维生素D受体作用蛋白205;PIBP:磷酸肌醇结合蛋白;SRC3/AIB1:类固醇受体辅激活因子3/乳腺癌扩增性抗原1;NCOA3:核受体辅激活子3;NRIF3:核受体作用因子3;ITGB3BP:整合素β3结合蛋白;ASC-1:激活信号辅整合子1;LZIP:亮氨酸拉链转录分子;CREB:环磷酸腺苷反应元件结合蛋白;PGC-1:过氧化物酶体增殖物激活受体γ辅激活因子1;TRBP:反式激活RNA结合蛋白;RAP250:核受体激活蛋白250;DRIP150:维生素D受体作用蛋白150;CIA:非依赖AF-2功能的辅激活因子;PNRC2:脯氨酸富集核受体辅激活因子2;TIP60:1型作用蛋白60;TRAP100:甲状腺激素受体相关蛋白100;PERC:过氧化物酶体增殖物激活受体辅激活因子;MNAR:雌激素受体非基因作用调节因子;PELP1:脯氨酸-谷氨酸-亮氨酸富集蛋白1;MLL2:混合谱系白血病基因编码蛋白2;CCDC 62:螺旋化螺旋结构域蛋白62;ERAP75:雌激素受体相关蛋白75;ACTN-4:辅肌动蛋白4;DAX-1:先天性肾上腺发育不良基因编码蛋白;SHP-1:包含SH-2功能域的蛋白酪氨酸磷酸酶1;RIP140:受体相互作用蛋白140;R-MGMT:DNA修复酶-甲基鸟嘌呤DNA甲基转移酶;LCoR:配体依赖的核受体辅抑制因子;REA:雌激素功能抑制因子;ERR10:雌激素相关受体10;NAG7:N-乙酰-β-D-氨基葡萄糖苷酶7;PROX1:Prospero同源盒基因1;ERK8:细胞外信号调节激酶8。 表 1 含有LXXLL模体的雌激素受体辅调节因子一览表
Table 1. Co-regulators containing LXXLL motif in estrogen receptor signal pathway
值得注意的是,虽然LXXLL模体在ER信号通路转导过程中起着重要作用,但并不代表LXXLL模体仅存在于ER或核受体信号途径中,很多其他蛋白质之间的相互作用也会涉及到LXXLL模体。有研究表明,LXXLL模体参与了慢性淋巴性白血病相关的基因转录沉默过程,且LXXLL模体在这一过程中的作用机制和其在核受体中的作用机制是不相同的[49]。另外,人们发现很多辅激活因子具有双重功能,它们一方面具有LXXLL模体,另一方面也具有结合LXXLL模体的结构域。例如,在核受体辅激活因子1中羧基端的PAS-B片段中就含有可与辅调节因子信号转导和转录激活子6中的LXXLL模体结合的区域,它们间的相互作用在白细胞介素4的功能中是不可缺少的[50]。最后,我们也应注意到很多ER辅调节因子虽然含有LXXLL模体,但其并不依赖LXXLL模体与ER的LBD结合[45, 49-51]。以上研究结果说明了LXXLL模体在细胞蛋白质-蛋白质相互作用中普遍存在,并且参与调节的过程十分复杂。
-
ER是核受体超家族中的一员,因此在结构和功能上,它与核受体超家族的其他成员具有很大的相似之处,随着核受体与LXXLL模体相互作用机制的研究不断取得进展,ER与LXXLL模体的相互作用机制也变得越来越清晰。多种核受体LBD的晶体结构分析揭示其存在一段保守的三维立体结构,它是一个三层反向平行的α螺旋三明治夹层结构,并在中间形成了疏水性的配体结合口袋[52-53]。如Feng等[54]通过基因点突变和X射线晶体衍射等手段发现甲状腺激素受体中存在一段区域,当它发生突变时,可以抑制甲状腺激素受体与辅激活因子糖皮质激素受体作用蛋白1的结合,但并不影响它同配体甲状腺激素的结合能力,预示着这一区域是与LXXLL模体结合所必需的。进一步研究发现此区域是由甲状腺激素受体LBD的helix-3、4、5和helix-12所组成,其中位于AF-2区域中的helix-12是高度活动的,当配体与LBD中的口袋结构结合后,它便被固定下来[54-55]。在其他核受体中,人们同样观察到了类似的结构,如在未与配体结合的视黄醇类X受体(RXR)结构中,LBD的helix-12是向外延伸游离的[56],当配体与其结合后,helix-12则贴紧LBD,并与配体直接接触[57-58]。
人们通过对大量核受体晶体结构的分析推断出,配体的结合可以诱导核受体中LBD构象的改变,这种改变涉及到helix-3、4、5及helix-12等构型的变化,结果暴露出一个疏水性的凹槽,凹槽两侧是由带电荷的氨基酸残基所组成的夹子结构(charge-clamp)。凹槽主要由LBD的helix-3和helix-12所维系,它就是配体依赖性的辅激活因子所结合的部位[54, 59]。
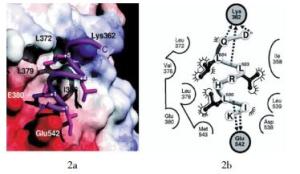
通过分析LBD与辅激活因子结合区域的晶体结构,显示LXXLL模体通过α螺旋构型的变化完成与LBD上AF-2表面的结合,在这一结合过程中,需要helix-3~5及helix-12的共同参与[57-58]。结合的最终结果是LXXLL模体上的3个亮氨酸被LBD上的疏水凹槽所包裹,其中+1和+5位置的亮氨酸嵌入疏水的凹槽中,+4位置的亮氨酸横跨在疏水槽的表面,而+2和+3位置的氨基酸残基则暴露在溶剂中,这一点和它们在不同核受体辅激活因子中的多变性是一致的。LXXLL模体与LBD的结合主要是通过疏水键和氢键这两种作用力实现的,它们都是特异性带电氨基酸残基之间的相互作用。如带电夹子(charge-clamp)分别通过helix-3上的亮氨酸残基和helix-12上的谷氨酸残基与LXXLL模体多肽骨干间发生氢键作用,这种作用力是十分牢固的[57, 59-60]。W?覿rnmark等[61]在研究辅调节因子转录中介因子2(transcriptional intermediary factor 2,TIF-2)与ER相互作用时,通过分析ER中LBD与TIF-2中LXXLL模体结合的晶体结构得到类似的结果(图 2),说明这一LXXLL模体结合模型在核受体信号中的普遍性。值得注意的是,虽然这一结合具有很高的亲和力,但LXXLL两侧的氨基酸残基仍会与核受体的LBD结合,由于这些氨基酸残基序列是各不相同的,因此,构成了不同辅激活因子与核受体结合的特异性[62-64]。
通过表 1我们可以发现很多辅调节因子中不止含有一个LXXLL模体,它们中的很多含有两个或多个LXXLL模体。有研究人员将SRC-1(含有5个LXXLL模体)切割成不同长度的小片段,发现相对于含有1个LXXLL模体的SRC-1片段,含有多个LXXLL模体的SRC-1片段可以以一种高度协调增强的方式与ER二聚体的LBD发生作用[65]。与此类似地,核受体因子过氧化物酶体增殖激活受体γ的同源二聚体可以和辅激活因子SRC-1上的多个LXXLL模体发生相互作用[60]。Rha等[66]在研究核受体肝细胞核因子4α(hepatocyte nuclear factor 4α,HNF4α)与辅激活因子PGC-1时也发现,PGC-1上的3个LXXLL序列可以随机地与HNF4α发生相互作用,而且随着LXXXL模体个数的增加,HNF4α的转录活性会随之增强。由此可见,辅调节因子上的LXXLL模体的个数与ER的转录活性具有一定的相关性,而且ER的二聚体可以同时与不同辅调节因子上的LXXLL模体发生相互作用。
另外,从表 1中我们还可以看到含有LXXLL模体的蛋白质既可以作为ER的辅激活因子,也可以作为辅抑制因子,这说明了其作用的复杂性。其中所涉及到的具体机制还不是很清楚,可能的解释是虽然这类辅抑制因子虽然也含有LXXLL模体,可以与LBD结合,但它们并不具备辅激活因子的相应酶活性或其他功能,从而阻断了辅激活因子与ER的结合,而一个核受体的辅调节因子包括多种,靶基因的最终转录活性取决于辅激活因子与辅抑制因子之间的平衡状态[3]。一般来说,核受体在缺乏配体的情况下,核受体辅抑制因子(nuclear receptor corepressor,NCoR)和维甲酸与甲状腺激素受体沉默体(silencing mediator for retinoid and thyroid hormone receptor,SMRT)等可以通过螺旋结构LXXI/HIXXXI/L模体与核受体的LBD结合,NCoR和SMRT等与LBD的结合位点和辅激活因子LXXLL模体结合的位点是同一部位,都是核受体上的疏水性的口袋结构,但是当兴奋性激素与核受体结合时,由于前者的亲和力较大,辅激活因子的LXXLL模体将取代辅抑制因子的LXXI/HIXXX/L模体,从而实现靶基因从阻遏状态向激活状态转变[45, 62]。但研究表明ER的抑制机制与其他核受体有着不同之处,在缺乏雌激素的情况下,ER不具备典型的转录抑制功能,只有当它结合到某些雌激素拮抗剂时,才显示出抑制功能[67-68]。它与SMRT、NCoR的结合也是比较独特的,SMRT、NCoR不仅可以与LBD的AF-2进行结合,也可以与ER的DNA结合区域发生相互作用[67-68]。因此,关于LXXLL模体与LXXI/HIXXXI/L模体之间的差异与联系以及它们所在ER辅调节因子中的功能和机制还有待进一步研究和证实。
-
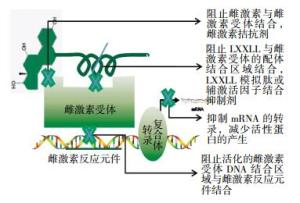
总的来说,涉及ER的药物设计思路可以分为4类,包括:①改变雌激素与ER的结合活性; ②阻碍已激活的ER与ERE的结合; ③抑制ER招募辅激活因子; ④抑制mRNA的转录与翻译活性(图 3)。传统的针对ER通路的药物主要集中在ER与配体间的结合位点上,人们据此研发了多种雌激素类似物,一般统称为选择性ER调节剂(selective estrogen receptor modulators,SERMs),临床上治疗雌激素阳性乳腺癌的常用药物他莫昔芬就属于SERMs类药物。这类药物的主要机制是可以模拟雌激素与ER的结合,但并不具备雌激素的生物活性,从而抑制ER的转录活性,最终有效地阻止雌激素依赖的肿瘤细胞的生长和增殖。然而,很多乳腺癌患者在长期使用SERMs后,会产生不同程度的耐药性,因此有必要研发新型药物,以作为SERMs的补充和替换[69]。
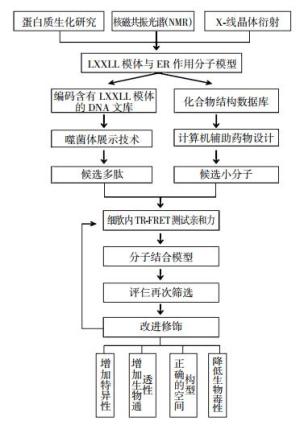
从上面的讨论中可以发现,辅激活因子的LXXLL模体与ER的结合位点具有很高的特异性,因此它们之间的相互作用被认为是一个非常有潜力的药物研发靶点。事实上,这一设想在很早就已经提出,人们也一直在尝试着研发这方面的药物(图 4)。概括地说,这些药物主要包括两大类,一类是非肽类小分子拮抗剂,又称辅激活因子结合抑制剂(coactivators binding inhibitors,CBIs),另一类主要是包含LXXLL模体的生物模拟肽(mimetics peptides)[64, 70]。
关于小分子拮抗剂的研究过程,一般研究人员首先分析LXXLL模体与ER结合的晶体结构,然后通过化合物结构数据库和计算机药物辅助设计等技术手段筛选出候选小分子,最后运用时间分辨-荧光共振能量转移技术(time-resolved fluorescence resonance energy transfer)进一步考察它们在细胞内与核受体的亲和力,以获得高亲和力特异性小分子。Rodriguez等[71]按照这种设计思路研发出两大类CIBs,第一类是根据由外向内的设计原理获得,包括多种杂环类化合物(三氮烯类、嘧啶类、三聚硫甲醛、环己烷类等),它们可以模拟LXXLL上的3个亮氨酸的结合作用; 而另一类是通过由内向外的途径获得的,主要是萘类化合物,它们可以模仿被嵌入的两个亮氨酸的作用,并通过向外延伸的取代基来模仿LXXLL模体两侧的其他基团。最后,通过分析各种CBIs与核受体上的LBD结合的特异性及强度,发现最有效的CBIs是嘧啶类家族[69, 71-72]。Gunther等[73]发现两性苯衍生物(amphipathic benzene scaffold)的疏水面可以模拟富含亮氨酸的α螺旋与ERα上凹槽之间的相互作用,且在体外试验中,只需要微摩尔级别的水平便可显著抑制ER的作用。之后研究人员陆续发现丙脒腙类化合物[74]、蜜蜂神经毒素(apamin)[75]、双环[2.2.2]辛烷(bicyclo[2.2.2]octanes)[76-77]等均有抑制LXXLL模体与ER结合的作用。
另外,在尝试运用含有LXXLL模体的小分子肽作为ER抑制剂方面,人们也付出了很多努力。这里最早的证据来自Mcinerney等[64]发现在经过转染质粒而高表达LXXLL多肽的细胞中,雌激素介导的转录活性是显著下降的。后来,研究人员运用噬菌体展示技术(phage display technology)初步筛选候选多肽,然后通过时间分辨-荧光共振能量转移技术评价其与核受体结合的亲和力与特异性[75]。虽然多肽类化合物十分容易通过生物化学途径获得,但这类小分子的缺点也是显而易见的。如在代谢过程中不稳定、容易被蛋白酶降解、很难穿透生物膜并准确地定位于作用位置、与靶分子作用时需要正确的空间构型等,这些也正是它们在临床实际应用中受限的主要原因[78]。因此,为了更好地利用这类小分子肽,尚需要大量工作来改善这些小分子的化学结构和生物学性能,如通过二硫键或硫醚键诱导LXXLL模体的螺旋构型的形成,采用非天然氨基酸(如叔亮氨酸、新戊基甘氨酸、青霉胺等)取代亮氨酸或半胱氨酸以进一步改善LXXLL的空间结构,添加聚乙二醇、亲脂类氨基酸等基团增加其生物膜通透性等。Galande等[69]综合运用这类手段获得了多种可以抑制辅激活因子与ER相互作用的小分子肽类,并称之为ER模拟肽调节剂(peptideomimetic estrogen-receptor modulators)。最近,Carraz等[79]采用人工合成的带有9个精氨酸尾部的LXXLL多肽(nona-arginine-LXXLL-peptide)直接处理细胞,结果发现此多肽可以渗透进入细胞并转位至细胞核,然后与辅激活因子相竞争,并可显著地抑制ERα介导的转录活性。
虽然针对LXXLL模体与ER结合位点的药物设计取得了很大的进展,但这一领域的研究还基本处于探索阶段,很多环节尚需要改进,如增加药物结构的稳定性与通透性、防止被机体降解、提高针对某一核受体作用的特异性、降低生物学毒性等。相信随着辅调节因子与ER作用机制的进一步明朗和模拟肽设计学的发展,这方面相关药物的问世只是时间问题。实际上,在核受体信号途径之外的模拟多肽的设计中,已经有了很多令人振奋的成果,如Moellering等[80]报道Notch转录因子复合体的抑制小分子多肽stapled-helical peptides derived from mastermind-like 1(SAHM1)可以抑制其转录活性复合体的形成,并在细胞和动物实验中均证实SAHM1具有良好的抗T细胞急性淋巴细胞白血病的作用。
-
鉴于ER在众多疾病形成发展中的重要作用,其相关研究一直是核受体信号研究领域的热点,并且已经在代谢性疾病、肿瘤的治疗和预防等方面取得了很多重要成果。从本文中我们可以发现,LXXLL模体广泛存在于核受体的辅调节因子中,这一点对于发现新的核受体辅调节因子具有重要的指导意义。同时考虑到LXXLL与ER LBD间结合的复杂性,有必要进一步研究它们之间的相互作用机制,尤其是针对LXXLL模体两侧序列的不确定性对LXXLL模体在ER中作用特异性的影响。此外,我们还需要进一步明确LXXLL模体在核受体复杂信号网络中的地位以及在其他蛋白质-蛋白质相互作用中所扮演的角色。毫无疑问,LXXLL模体生化结构特征的相关研究成果也将为它的应用提供新的契机和机遇。Hu等[81]研究发现B细胞迁移基因/erbB2转录因子抗增殖蛋白质家族含有LXXLL序列,并发现其中B细胞迁移基因1可以通过ER途径提高非小细胞肺癌细胞的放射敏感性[82],我们将进一步明确B细胞迁移基因/erbB2转录因子抗增殖蛋白质家族在核受体信号通路中所发挥的作用,并研究其与雌激素、SERMs、放化疗之间的相互影响,希望能发现激素阳性乳腺癌新的治疗靶点。
LXXLL模体在雌激素受体信号通路中的作用及应用价值
Role of LXXLL motif in modulation of estrogen receptor signaling and its potential application
-
摘要: 雌激素受体是核受体超家族中重要的一员,其与雌激素的结合在人类组织器官发育、新陈代谢以及许多疾病的发生和发展中起着重要的作用。协同调节因子是雌激素-雌激素受体信号转录传导途径中的一类重要调节分子,它们可以与雌激素受体直接结合,并作为协同激活因子上调或作为协同抑制因子降低雌激素受体的基因转录功能。蛋白分子结构分析发现,在协同调节因子与雌激素受体的相互作用中,其蛋白序列中含有的一段非常保守的富含亮氨酸的螺旋序列LXXLL模序(L指亮氨酸、X指任意氨基酸)扮演着重要角色,并在辅调节因子,特别是在协同激活因子中广泛地存在。深入研究LXXLL模序与雌激素受体相互作用的分子机制不仅有助于对LXXLL模序在雌激素受体-辅助调节因子蛋白之间相互作用中地位的理解,也为针对LXXLL模体与核受体的作用位点设计新的靶向药物奠定了基础。因此,该文简述了LXXLL模序在雌激素受体协同调节因子中存在的广泛性、LXXLL模序与雌激素受体相互作用的机制以及其在新药设计上的应用等相关方面的研究进展。
-
关键词:
- 受体,雌激素 /
- 协同调节因子 /
- LXXLL模序 /
- 共激活因子结合抑制剂 /
- 模拟肽
Abstract: Estrogen receptor(ER), one member of the nuclear receptor superfamily, via binding to estrogen, plays important roles in development and metabolism of human tissues and organs as well as a number of diseases, such as cancer, obesity, diabetes and osteoporosis. Co-regulators, one important kind of factors in the ER transcriptional signaling, increase(as co-activators) or reduce(as co-repressors)ER-mediated transcriptional activity via direct and indirect binding to ER. Increasing evidence has revealed that a short α-helical sequence LXXLL motif(where L is leucine, X is any amino acid), widely present in many co-regulators, especially co-activators, plays an essential or necessary role in co-regulator interaction with ER. To explore the involved mechanism(s) will be helpful in understanding the critical role of LXXLL motif in the interaction of co-regulators and ER. This review briefly describes the potential roles of LXXLL motif in the involvement of ER co-regulators in modulation of ER signaling and underlying mechanisms as well as current progress in developing pharmaceutical agents that modulate ER-coregulator interaction via specifically targeting LXXLL motifs in therapy of hormone-associated cancers.-
Key words:
- Receptors, estrogen /
- Co-regulators /
- LXXLL motif /
- Co-activator binding inhibitors /
- Mimetic Peptide
-
表 1 含有LXXLL模体的雌激素受体辅调节因子一览表
Table 1. Co-regulators containing LXXLL motif in estrogen receptor signal pathway
基因 人类基因编号 LXXLL序列(氨基酸位点) 在核受体中的作用 蛋白质功能 在疾病发生中的作用 发现时间 参考文献 CBP 1387 QLSELLRG(68-75)
QLVLLLHA(356-363)辅激活因子 组蛋白乙酰化,与SRC家族相互作用 在亨廷顿舞蹈症、白血病、脊延髓肌肉萎缩中异常表达 1996 [9] p300 2033 KLSELLRS(80-87)
QLVLLLHA(341-348)
ALQNLLRT(2050-1057)辅激活因子 组蛋白乙酰化,与SRC家族相互作用 在胃肠道肿瘤中杂合性缺失、白血病中形成异常Moz融合蛋白,突变可引起多发性肿瘤 1996 [10] SRC-1/NCOA1 8648 KLVQLLTT(252-260)
ILHRLLQE(309-318)
LLRYLLDK(368-375)
QLDELLCP(532-539)
LLQQLLTE(1054-1061)辅激活因子 组蛋白乙酰化,参与信号转导和转录激活子家族介导的转录激活 在乳腺癌、前列腺癌、子宫内膜癌中过量表达,突变可引起横纹肌肉瘤 1997 [7] SRC2/GRIP1/TIF2 10499 KLLQLLTT(640-647)
ILHRLLQD(689-696)
LLRYLLDK(744-751)
QLGRLLPN(877-884)辅激活因子 组蛋白乙酰化、与辅激活因子相关精氨酸甲基转移酶1和EP300/p300等形成复合物 在乳腺癌、脑部肿瘤中过量表达,在白血病中形成Moz-tif融合蛋白 1998 [11-13] pCAF 8850 YLFGLLRK(189-196) 辅激活因子 组蛋白乙酰化,与雌激素受体的DNA结合区域相互作用 缺失可导致直结肠癌 1998 [14-15] TRAP220/MED1/DRIP205/PIBP 5469 ILTSLLQI(603-610)
MLMNLLKD(644-651)辅激活因子 中介体,与众多MEDs形成调节复合体 缺失可导致Williams综合症 1998 [16-17] SRC3/AIB1/NCOA3 8202 KLVQLLTT(621-628)
ILHRLLQD(684-691)
LLRYLLDK(735-742)辅激活因子 组蛋白乙酰化,可与CBP/p300形成激活复合物 基因变异可导致乳腺癌、卵巢癌,在直结肠癌中过量表达 1998 [18-19] NRIF3/ITGB3BP 23421 FLKALLN(8-15)
KLDGLLEE(171-177)辅激活因子 与蛋氨酸腺苷转移酶1相互作用,调节乳腺癌细胞对雌激素的敏感程度 有待研究 1999 [20-21] ASC-1 9325 RLAVLLPG(160-167) 辅激活因子 招募组蛋白乙酰化酶,与CBP/p00、SRC-1相互作用,在核转录因子κB、激活蛋白1的转录激活中发挥重要作用 有待研究 1999 [22] LZIP/CREB3 17039 DLLALLEE(12-19)
DLLSLLSP(52-59)辅激活因子 可以与环磷酸腺苷反应元件结合,调控细胞增殖,可正向调控核转录因子κB通路,与疱疹病毒VP16感染有联系 增加白血病的迁移 2000 [23] PGC-1 10891 LLKKLLLA(143-147) 辅激活因子 参与mRNA的剪切,正向调节组蛋白乙酰化,并在能量代谢和脂肪细胞分化中具有重要作用 表达水平与乳腺癌的病死率成反比,且在心血管和糖尿病等疾病中异常表达 2000 [24-25] TRBP/RAP250/ASC-2 23054 LLVNLLQS(886-893)
LLSQLLDN(1490-1497)辅激活因子 招募组蛋白乙酰化酶,并参与DNA复制和损伤修复 突变可导致乳腺癌 2000 [26-27] DRIP150/MED14 9282 ALNILLLP(69-76)
VLTDLLPR(1181-1188)辅激活因子 中介体,与众多MEDs形成调节复合体,参与RNA聚合酶II的转录功能 有待研究 2000 [28] CIA 57727 SLINLLAD(386-393) 辅激活因子/辅抑制因子 具备氨酰基-转运RNA连接酶功能,可以与ATP结合,且与雌激素受体的结合不依赖于转录激活区2 有待研究 2001 [29] PNRC2 55629 QLKTLLKV(171-177) 辅激活因子 参与无意义密码子介导的mRNA降解 有待研究 2001 [30] TIP60 10524 MLKRLLRI(492-499) 辅激活因子 组蛋白乙酰化酶复合体NuA4的催化成分,且可以直接使毛细血管扩张共济失调突变基因乙酰化,在细胞修复中发挥作用 有待研究 2001 [31] PERC/PGC-1 133522 LLQKLLLA(155-162)
ILRELLAQ(342-349)
ILRELLAQ(342-349)辅激活因子 转录调节复合体成分之一,可以与RNA结合,参与能量代谢调节和脂肪细胞分化 基因突变可导致乳腺癌、肥胖、2型糖尿病,在甲状腺嗜酸细胞瘤中过量表达 2002 [32-33] TRAP100/MED24 9862 SLDLLLAA(447-454)
ILPGLLTD(787-794)辅激活因子 中介体,与众多MEDs形成调节复合体,参与RNA聚合酶II的转录功能 在乳腺癌中过量表达 2002 [34-35] MNAR/PELP1 27043 CLLSLLYG(28-35)
VLRDLLRY(72-79)
HLPGLLTS(94-101)
LLTSLLGL(98-105)
ELHSLLAS(181-188)
SLHTLLGA(188-195)
PLRLLLLP(281-289)
LLTHLLSD(376-383)
ELYCLLLA(496-503)
LLALLLAP(501-508)辅激活因子 可以与生长因子信号通路中组分表皮生长因子受体、肝细胞生长因子调节酪氨酸激酶作用物等相互作用,通过视网膜母细胞瘤抑制蛋白1在雌激素介导的细胞周期进展中发挥作用,并且同SRC、磷脂酰肌醇3激酶、信号转导和转录激活子3等相互作用 细胞质内浓度增高时可以导致乳腺癌,过量表达可导致子宫内膜癌、涎腺导管癌等 2004 [36-37] MLL2 8055 RLRELLIR(2685-2691)
HLDDLLNG(3037-3043)
QLQALLMQ(4221-4227)
PLOGLLGC(4252-4258)
LLQKLLRA(4187-4194)
RLRLLLTT(4714-4721)辅激活因子 具有甲基转移酶的功能,可以使组蛋白H3的Lys-3甲基化,被雌激素受体1所招募激活雌激素受体功能 缺失可导致Kabuki综合征,在乳腺癌中过量表达 2006 [38-39] CCDC62/ERAP75 84600 KLQRLLAE(633-640)
ELSTLLPI(649-656)辅激活因子 可以增强雌激素介导的cyclinD1的表达和促进细胞的生长 在前列腺癌细胞中广泛表达 2009 [40] ACTN-4 43707 KLMLLLEV(83-90) 辅激活因子 肌动蛋白结合蛋白,苯丙胺调节转录因子复合物的组成部分 突变可导致1型局灶节段性肾小球硬化 2011 [41] DAX-1 190 ILYSLLTS(145-152) 辅抑制因子 可与核受体亚家族5-A组-成员1蛋白和核受体亚家族0-B组-成员2蛋白形成多异聚体,并招募其他辅抑制子,在肾上腺激素轴的发育中具有重要作用 缺失可导致X染色体偶联的阿狄森氏病和剂量敏感性性反转 2000 [8] SHP-1 5777 ILYALLTS(19-26) 辅抑制因子 可以与DNA直接结合 突变与早发性肥胖有关 2000 [8] RIP140 8204 YLEGLLMH(20-27)
LLASLLQS(132-139)
HLKTLLKK(184-191)
QLALLLSS(265-272)
LLLHLLKS(379-386)
LLLLLLGH(499-506)
VLQLLLGN(712-719)
LLSRLLRQ(818-825)
VLKQLLLS(935-972)辅抑制因子 招募组蛋白去乙酰化酶1、组蛋白去乙酰化酶3等组蛋白去乙酰化酶,并与核受体辅激活子1形成复合物 突变可导致子宫内膜癌和不孕 2001 [42] R-MGMT 4255 VLWKLLKV(95-105) 辅抑制因子 具有甲基转移酶的活性,在DNA损伤后,可以抑制雌激素受体介导的细胞增殖 基因突变或单倍体可导致膀胱癌和脑部肿瘤,在肺癌中过量表达 2001 [43] LCoR 84458 VLSKLLMA(52-59) 辅抑制因子 可以与DNA直接结合,招募羧基末端结合蛋白1、组蛋白去乙酰化酶3、组蛋白去乙酰化酶6等共同组成转录辅抑制复合物 有待研究 2003 [44] REA 11331 ALKLLLGA(22-29) 辅抑制因子 中介体,招募组蛋白去乙酰化酶 在乳腺癌中低表达 2003 [45] ERR10/NAG7 29931 FLIWLLCW(336-343)
FLPHLLNQ(322-329)辅抑制因子 抑制雌激素受体调c-Jun氨基末端激酶琢的活性/激活蛋白,并上1/金属基质蛋白酶1信号通路 在胃肠肿瘤中高表达,可以增强鼻咽癌的侵袭能力 2004 [46] PROX1 5629 VLRKLLKR(69-76) 辅抑制因子 在机体的神经系统和内脏发育中发挥重要作用 低表达可导致肝癌、胰腺癌等 2006 [47] ERK8 9606 TLDALLPP(264-271)
LLRRLLVF(280-287)辅抑制因子 具备ATP结合活性和苏氨酸/丝氨酸激酶的活性,参与蛋白的磷酸化 有待研究 2011 [48] 注:CBP:CREB结合蛋白;SRC:类固醇受体辅激活因子;SRC-1/NCOA-1:类固醇/核受体辅激活因子1;SRC2/GRIP1:类固醇受体激活子2/糖皮质激素受体作用蛋白1;PCAF:P300/CBP相关因子;TRAP220:甲状腺激素受体相关蛋白220;MED:甲基化CpG结合核酸内切酶;DRIP205:维生素D受体作用蛋白205;PIBP:磷酸肌醇结合蛋白;SRC3/AIB1:类固醇受体辅激活因子3/乳腺癌扩增性抗原1;NCOA3:核受体辅激活子3;NRIF3:核受体作用因子3;ITGB3BP:整合素β3结合蛋白;ASC-1:激活信号辅整合子1;LZIP:亮氨酸拉链转录分子;CREB:环磷酸腺苷反应元件结合蛋白;PGC-1:过氧化物酶体增殖物激活受体γ辅激活因子1;TRBP:反式激活RNA结合蛋白;RAP250:核受体激活蛋白250;DRIP150:维生素D受体作用蛋白150;CIA:非依赖AF-2功能的辅激活因子;PNRC2:脯氨酸富集核受体辅激活因子2;TIP60:1型作用蛋白60;TRAP100:甲状腺激素受体相关蛋白100;PERC:过氧化物酶体增殖物激活受体辅激活因子;MNAR:雌激素受体非基因作用调节因子;PELP1:脯氨酸-谷氨酸-亮氨酸富集蛋白1;MLL2:混合谱系白血病基因编码蛋白2;CCDC 62:螺旋化螺旋结构域蛋白62;ERAP75:雌激素受体相关蛋白75;ACTN-4:辅肌动蛋白4;DAX-1:先天性肾上腺发育不良基因编码蛋白;SHP-1:包含SH-2功能域的蛋白酪氨酸磷酸酶1;RIP140:受体相互作用蛋白140;R-MGMT:DNA修复酶-甲基鸟嘌呤DNA甲基转移酶;LCoR:配体依赖的核受体辅抑制因子;REA:雌激素功能抑制因子;ERR10:雌激素相关受体10;NAG7:N-乙酰-β-D-氨基葡萄糖苷酶7;PROX1:Prospero同源盒基因1;ERK8:细胞外信号调节激酶8。 -
[1] Deroo BJ, Korach KS. Estrogen receptors and human disease[J]. J Clin Invest, 2006, 116(3):561-570. doi: 10.1172/JCI27987 [2] Leung YK, Mak P, Hassan S, et al. Estrogen receptor(ER)-beta isoforms:a key to understanding ER-beta signaling[J]. Proc Natl Acad Sci U S A, 2006, 103(35):13162-13167. [3] Heldring N, Pike A, Andersson S, et al. Estrogen receptors:how do they signal and what are their targets[J]. Physiol Rev, 2007, 87(3):905-931. [4] Li X, Huang J, Yi P, et al. Single-chain estrogen receptors(ERs) reveal that the ERalpha/beta heterodimer emulates functions of the ERalpha dimer in genomic estrogen signaling pathways[J]. Mol Cell Biol, 2004, 24(17):7681-7694. [5] Aranda A, Pascual A. Nuclear hormone receptors and gene expression[J]. Physiol Rev, 2001, 81(3):1269-1304. doi: 10.1152/physrev.2001.81.3.1269 [6] Glass CK, Rosenfeld MG. The coregulator exchange in transcriptional functions of nuclear receptors[J]. Genes Dev, 2000, 14(2):121-141. [7] Heery DM, Kalkhoven E, Hoare S, et al. A signature motif in transcriptional co-activators mediates binding to nuclear receptors[J]. Nature, 1997, 387(6634):733-736. [8] Zhang H, Thomsen JS, Johansson L, et al. DAX-1 functions as an LXXLL-containing corepressor for activated estrogen receptors[J]. J Biol Chem, 2000, 275(51):39855-39859. [9] Ogryzko VV, Schiltz RL, Russanova V, et al. The transcriptional coactivators p300 and CBP are histone acetyltransferases[J]. Cell, 1996, 87(5):953-959. [10] Koshiishi N, Chong JM, Fukasawa T, et al. p300 gene alterations in intestinal and diffuse types of gastric carcinoma[J]. Gastric Cancer, 2004, 7(2):85-90. [11] Ding XF, Anderson CM, Ma H, et al. Nuclear receptor-binding sites of coactivators glucocorticoid receptor interacting protein 1(GRIP1)and steroid receptor coactivator 1(SRC-1):multiple motifs with different binding specificities[J]. Mol Endocrinol, 1998, 12(2):302-313. [12] Voegel JJ, Heine MJ, Tini M, et al. The coactivator TIF2 contains three nuclear receptor-binding motifs and mediates transactivation through CBP binding-dependent and -independent pathways[J]. EMBO J, 1998, 17(2):507-519. [13] Kurebayashi J, Otsuki T, Kunisue H, et al. Expression levels of estrogen receptor-alpha, estrogen receptor-beta, coactivators, and corepressors in breast cancer[J]. Clin Cancer Res, 2000, 6(2):512-518. [14] Blanco JC, Minucci S, Lu J, et al. The histone acetylase PCAF is a nuclear receptor coactivator[J]. Genes Dev, 1998, 12(11):1638-1651. [15] Giannini R, Cavallini A. Expression analysis of a subset of coregulators and three nuclear receptors in human colorectal carcinoma[J]. Anticancer Res, 2005, 25(6B):4287-4292. [16] Yuan CX, Ito M, Fondell JD, et al. The TRAP220 component of a thyroid hormone receptor-associated protein(TRAP) coactivator complex interacts directly with nuclear receptors in a ligand-dependent fashion[J]. Proc Natl Acad Sci U S A, 1998, 95(14):7939-7944. [17] Kitagawa H, Fujiki R, Yoshimura K, et al. The chromatin-remodeling complex WINAC targets a nuclear receptor to promoters and is impaired in Williams syndrome[J]. Cell, 2003, 113(7):905-917. [18] Li H, Chen JD. The receptor-associated coactivator 3 activates transcription through CREB-binding protein recruitment and autoregulation[J]. J Biol Chem, 1998, 273(10):5948-5954. [19] Anzick SL, Kononen J, Walker RL, et al. AIB1, a steroid receptor coactivator amplified in breast and ovarian cancer[J]. Science, 1997, 277(5328):965-968. [20] Li D, Desai-Yajnik V, Lo E, et al. NRIF3 is a novel coactivator mediating functional specificity of nuclear hormone receptors[J]. Mol Cell Biol, 1999, 19(10):7191-7202. [21] Talukder AH, Gururaj A, Mishra SK, et al. Metastasis-associated protein 1 interacts with NRIF3, an estrogen-inducible nuclear receptor coregulator[J]. Mol Cell Biol, 2004, 24(15):6581-6591. [22] Kim HJ, Yi JY, Sung HS, et al. Activating signal cointegrator 1, a novel transcription coactivator of nuclear receptors, and its cytosolic localization under conditions of serum deprivation[J]. Mol Cell Biol, 1999, 19(9):6323-6332. [23] Luciano RL, Wilson AC. N-terminal transcriptional activation domain of LZIP comprises two LxxLL motifs and the host cell factor-1 binding motif[J]. Proc Natl Acad Sci U S A, 2000, 97(20):10757-10762. [24] Vega RB, Huss JM, Kelly DP. The coactivator PGC-1 cooperates with peroxisome proliferator-activated receptor alpha in transcriptional control of nuclear genes encoding mitochondrial fatty acid oxidation enzymes[J]. Mol Cell Biol, 2000, 20(5):1868-1876. [25] Puigserver P, Spiegelman BM. Peroxisome proliferator-activated receptor-gamma coactivator 1 alpha(PGC-1 alpha):transcriptional coactivator and metabolic regulator[J]. Endocr Rev, 2003, 24(1):78-90. [26] Zhu Y, Kan L, Qi C, et al. Isolation and characterization of peroxisome proliferator-activated receptor(PPAR)interacting protein(PRIP)as a coactivator for PPAR[J]. J Biol Chem, 2000, 275(18):13510-13516. [27] Sjöblom T, Jones S, Wood LD, et al. The consensus coding sequences of human breast and colorectal cancers[J]. Science, 2006, 314(5797):268-274. [28] Ko L, Cardona GR, Chin WW. Thyroid hormone receptor-binding protein, an LXXLL motif-containing protein, functions as a general coactivator[J]. Proc Natl Acad Sci U S A, 2000, 97(11):6212-6217. [29] Sauvé F, McBroom LD, Gallant J, et al. CIA, a novel estrogen receptor coactivator with a bifunctional nuclear receptor interacting determinant[J]. Mol Cell Biol, 2001, 21(1):343-353. [30] Zhou D, Chen S. PNRC2 is a 16 kDa coactivator that interacts with nuclear receptors through an SH3-binding motif[J]. Nucleic Acids Res, 2001, 29(19):3939-3948. [31] Gaughan L, Brady ME, Cook S, et al. Tip60 is a co-activator specific for class I nuclear hormone receptors[J]. J Biol Chem, 2001, 276(50):46841-46848. [32] Kressler D, Schreiber SN, Knutti D, et al. The PGC-1-related protein PERC is a selective coactivator of estrogen receptor alpha[J]. J Biol Chem, 2002, 277(16):13918-13925. [33] Meirhaeghe A, Crowley V, Lenaghan C, et al. Characterization of the human, mouse and rat PGC1 beta(peroxisome-proliferator-activated receptor-gamma co-activator 1 beta)gene in vitro and in vivo[J]. Biochem J, 2003, 373(Pt 1):155-165. [34] Wang Q, Sharma D, Ren Y, et al. A coregulatory role for the TRAP-mediator complex in androgen receptor-mediated gene expression[J]. J Biol Chem, 2002, 277(45):42852-42858. [35] Clark J, Edwards S, John M, et al. Identification of amplified and expressed genes in breast cancer by comparative hybridization onto microarrays of randomly selected cDNA clones[J]. Genes Chromosomes Cancer, 2002, 34(1):104-114. [36] Barletta F, Wong CW, Mcnally C, et al. Characterization of the interactions of estrogen receptor and MNAR in the activation of cSrc[J]. Mol Endocrinol, 2004, 18(5):1096-1108. [37] Vadlamudi RK, Manavathi B, Balasenthil S, et al. Functional implications of altered subcellular localization of PELP1 in breast cancer cells[J]. Cancer Res, 2005, 65(17):7724-7732. [38] Mo R, Rao SM, Zhu YJ. Identification of the MLL2 complex as a coactivator for estrogen receptor alpha[J]. J Biol Chem, 2006, 281(23):15714-15720. [39] Choudhary C, Kumar C, Gnad F, et al. Lysine acetylation targets protein complexes and co-regulates major cellular functions[J]. Science, 2009, 325(5942):834-840. [40] Chen M, Ni J, Chang HC, et al. CCDC62/ERAP75 functions as a coactivator to enhance estrogen receptor beta-mediated transactivation and target gene expression in prostate cancer cells[J]. Carcinogenesis, 2009, 30(5):841-850. [41] Khurana S, Chakraborty S, Cheng XW, et al. The actin-binding protein, actinin alpha 4(ACTN4), is a nuclear receptor coactivator that promotes proliferation of MCF-7 breast cancer cells[J]. J Biol Chem, 2011, 286(3):1850-1859. [42] Heery DM, Hoare S, Hussain S, et al. Core LXXLL motif sequences in CREB-binding protein, SRC1, and RIP140 define affinity and selectivity for steroid and retinoid receptors[J]. J Biol Chem, 2001, 276(9):6695-6702. [43] Teo AK, Oh HK, Ali RB, et al. The modified human DNA repair enzyme O(6)-methylguanine-DNA methyltransferase is a negative regulator of estrogen receptor-mediated transcription upon alkylation DNA damage[J]. Mol Cell Biol, 2001, 21(20):7105-7114. doi: 10.1128/MCB.21.20.7105-7114.2001 [44] Fernandes I, Bastien Y, Wai T, et al. Ligand-dependent nuclear receptor corepressor LCoR functions by histone deacetylase-dependent and -independent mechanisms[J]. Mol Cell, 2003, 11(1):139-150. doi: 10.1016/S1097-2765(03)00014-5 [45] Martini PG, Katzenellenbogen BS. Modulation of estrogen receptor activity by selective coregulators[J]. J Steroid Biochem Mol Biol, 2003, 85(2-5):117-122. [46] Meng Q, Zhao Z, Yan M, et al. ERR-10:a new repressor in transcriptional signaling activation of estrogen receptor-alpha[J]. FEBS Lett, 2004, 576(1-2):190-200. [47] Song KH, Li T, Chiang JY. A Prospero-related homeodomain protein is a novel co-regulator of hepatocyte nuclear factor 4alpha that regulates the cholesterol 7alpha-hydroxylase gene[J]. J Biol Chem, 2006, 281(15):10081-10088. [48] Rossi M, Colecchia D, Iavarone C, et al. Extracellular signal-regulated kinase 8(ERK8) controls estrogen-related receptor α(ERRα)cellular localization and inhibits its transcriptional activity[J]. J Biol Chem, 2011, 286(10):8507-8522. [49] Zhang J, Kalkum M, Yamamura S, et al. E protein silencing by the leukemogenic AML1-ETO fusion protein[J]. Science, 2004, 305(5688):1286-1289. [50] Razeto A, Ramakrishnan V, Litterst CM, et al. Structure of the NCoA-1/SRC-1 PAS-B domain bound to the LXXLL motif of the STAT6 transactivation domain[J]. J Mol Biol, 2004, 336(2):319-329. [51] Benecke A, Gaudon C, Garnier J M, et al. ADA3-containing complexes associate with estrogen receptor alpha[J]. Nucleic Acids Res, 2002, 30(11):2508-2514. [52] Bourguet W, Germain P, Gronemeyer H. Nuclear receptor ligand-binding domains:three-dimensional structures, molecular interactions and pharmacological implications[J]. Trends Pharmacol Sci, 2000, 21(10):381-388. [53] Moras D, Gronemeyer H. The nuclear receptor ligand-binding domain:structure and function[J]. Curr Opin Cell Biol, 1998, 10(3):384-391. [54] Feng W, Ribeiro RC, Wagner RL, et al. Hormone-dependent coactivator binding to a hydrophobic cleft on nuclear receptors[J]. Science, 1998, 280(5370):1747-1749. [55] Baniahmad A, Leng X, Burris TP, et al. The tau 4 activation domain of the thyroid hormone receptor is required for release of a putative corepressor(s)necessary for transcriptional silencing[J]. Mol Cell Biol, 1995, 15(1):76-86. [56] Bourguet W, Ruff M, Chambon P, et al. Crystal structure of the ligand-binding domain of the human nuclear receptor RXR-alpha[J]. Nature, 1995, 375(6530):377-382. [57] Shiau AK, Barstad D, Loria PM, et al. The structural basis of estrogen receptor/coactivator recognition and the antagonism of this interaction by tamoxifen[J]. Cell, 1998, 95(7):927-937. [58] Brzozowski AM, Pike AC, Dauter Z, et al. Molecular basis of agonism and antagonism in the oestrogen receptor[J]. Nature, 1997, 389(6652):753-758. [59] Darimont BD, Wagner RL, Apriletti JW, et al. Structure and specificity of nuclear receptor-coactivator interactions[J]. Genes Dev, 1998, 12(21):3343-3356. [60] Nolte RT, Wisely GB, Westin S, et al. Ligand binding and co-activator assembly of the peroxisome proliferator-activated receptor-gamma[J]. Nature, 1998, 395(6698):137-143. [61] Wärnmark A, Treuter E, Gustafsson JA, et al. Interaction of transcriptional intermediary factor 2 nuclear receptor box peptides with the coactivator binding site of estrogen receptor alpha[J]. J Biol Chem, 2002, 277(24):21862-21868. [62] Klein F, Atkinson RA, Potier N, et al. Biochemical and NMR mapping of the interface between CREB-binding protein and ligand binding domains of nuclear receptor-Beyond the LXXLL motif[J]. J Biol Chem, 2005, 280(7):5682-5692. [63] Hur E, Pfaff SJ, Payne ES, et al. Recognition and accommodation at the androgen receptor coactivator binding interface[J]. PLoS Biol, 2004, 2(9):E274. [64] Mcinerney EM, Rose DW, Flynn SE, et al. Determinants of coactivator LXXLL motif specificity in nuclear receptor transcriptional activation[J]. Genes Dev, 1998, 12(21):3357-3368. [65] Gee AC, Carlson KE, Martini PG, et al. Coactivator peptides have a differential stabilizing effect on the binding of estrogens and antiestrogens with the estrogen receptor[J]. Mol Endocrinol, 1999, 13(11):1912-1923. [66] Rha GB, Wu G, Shoelson SE, et al. Multiple binding modes between HNF4alpha and the LXXLL motifs of PGC-1alpha lead to full activation[J]. J Biol Chem, 2009, 284(50):35165-35176. [67] Varlakhanova N, Snyder C, Jose S, et al. Estrogen receptors recruit SMRT and N-CoR corepressors through newly recognized contacts between the corepressor N terminus and the receptor DNA binding domain[J]. Mol Cell Biol, 2010, 30(6):1434-1445. [68] Heldring N, Pawson T, Mcdonnell D, et al. Structural insights into corepressor recognition by antagonist-bound estrogen receptors[J]. J Biol Chem, 2007, 282(14):10449-10455. [69] Galande AK, Bramlett KS, Trent JO, et al. Potent inhibitors of LXXLL-based protein-protein interactions[J]. Chembiochem, 2005, 6(11):1991-1998. [70] Chang Cy, Norris JD, Grøn H, et al. Dissection of the LXXLL nuclear receptor-coactivator interaction motif using combinatorial peptide libraries:discovery of peptide antagonists of estrogen receptors alpha and beta[J]. Mol Cell Biol, 1999, 19(12):8226-8239. [71] Rodriguez AL, Tamrazi A, Collins ML, et al. Design, synthesis, and in vitro biological evaluation of small molecule inhibitors of estrogen receptor alpha coactivator binding[J]. J Med Chem, 2004, 47(3):600-611. [72] Parent AA, Gunther JR, Katzenellenbogen JA. Blocking estrogen signaling after the hormone:pyrimidine-core inhibitors of estrogen receptor-coactivator binding[J]. J Med Chem, 2008, 51(20):6512-6530. [73] Gunther JR, Moore TW, Collins ML, et al. Amphipathic benzenes are designed inhibitors of the estrogen receptor alpha/steroid receptor coactivator interaction[J]. ACS Chem Biol, 2008, 3(5):282-286. [74] Lafrate AL, Gunther JR, Carlson KE, et al. Synthesis and biological evaluation of guanylhydrazone coactivator binding inhibitors for the estrogen receptor[J]. Bioorg Med Chem, 2008, 16(23):10075-10084. [75] Phan T, Nguyen HD, Göksel H, et al. Phage display selection of miniprotein binders of the estrogen receptor[J]. Chem Commun(Camb), 2010, 46(43):8207-8209. [76] Shao D, Berrodin TJ, Manas E, et al. Identification of novel estrogen receptor alpha antagonists[J]. J Steroid Biochem Mol Biol, 2004, 88(4-5):351-360. [77] Zhou HB, Collins ML, Gunther JR, et al. Bicyclo[2. 2. 2]octanes:close structural mimics of the nuclear receptor-binding motif of steroid receptor coactivators[J]. Bioorg Med Chem Lett, 2007, 17(15):4118-4122. [78] Hruby VJ, Li G, Haskell-Luevano C, et al. Design of peptides, proteins, and peptidomimetics in chi space[J]. Biopolymers, 1997, 43(3):219-266. [79] Carraz M, Zwart W, Phan T, et al. Perturbation of estrogen receptor alpha localization with synthetic nona-arginine LXXLL-peptide coactivator binding inhibitors[J]. Chem Biol, 2009, 16(7):702-711. [80] Moellering RE, Cornejo M, Davis TN, et al. Direct inhibition of the NOTCH transcription factor complex[J]. Nature, 2009, 462(7270):182-188. [81] Hu XD, Meng QH, Xu JY, et al. BTG2 is an LXXLL-dependent co-repressor for androgen receptor transcriptional activity[J]. Biochem Biophys Res Commun, 2011, 404(4):903-909. [82] Sun KK, Zhong N, Yang Y, et al. Enhanced radiosensitivity of NSCLC cells by transducer of erbB2. 1(TOB1)through modulation of the MAPK/ERK pathway[J]. Oncol Rep, 2013, 29(6):2385-2391. -




 下载:
下载: