-
电离辐射(ionizing radiation,IR)通过直接或间接方式对DNA造成损伤,其可能导致基因突变、染色体畸变、细胞癌变等不利的生物学效应,因此DNA损伤及其修复机制一直是放射生物学领域研究的重点问题。蛋白质翻译后修饰(post-translational modification,PTM)是蛋白质被翻译出来后所经历的一个共价加工过程,即通过在氨基酸侧链上共价结合化学小分子基团或其他多肽,或通过蛋白质水解剪去基团从而使蛋白质的性质发生改变。泛素化修饰和小泛素样修饰物(small ubiquitin-like modifier,SUMO)化修饰作为PTM的2种类型,在IR诱导的DNA损伤修复中发挥着重要作用[1]。本文对泛素化修饰和SUMO化修饰在DNA损伤修复中的作用进行综述,为深入了解IR诱导的DNA损伤修复机制提供参考。
-
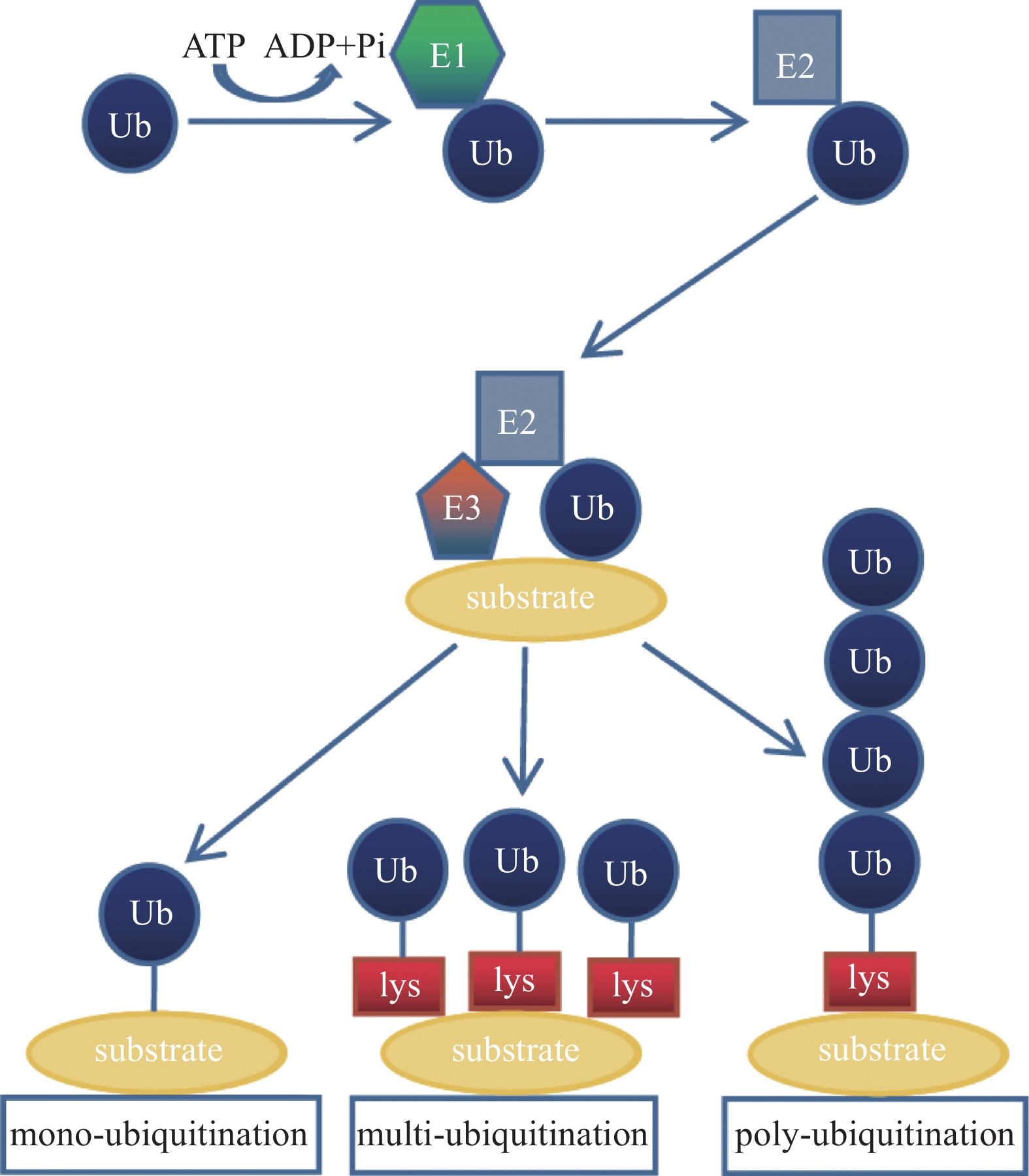
泛素由76个氨基酸组成,分子量约为8 451 Da,是一种存在于大部分真核细胞中、进化过程高度保守的小分子蛋白质。其全长包含7个赖氨酸位点(K6、K11、K27、K29、K33、K48和K63)、1个位于N端的甲硫氨酸位点(M1)以及1个位于C端的甘氨酸位点(G76)[2]。不同的赖氨酸位点与不同类型的E3泛素连接酶相互作用,形成复杂的泛素化修饰,在细胞周期、DNA修复、蛋白质降解等过程中发挥重要作用。根据目标蛋白上的泛素数量和位置,可以将其分为单泛素化修饰、多泛素化修饰和多聚泛素化修饰。单泛素化修饰是目标蛋白结合单个泛素分子;多泛素化修饰是目标蛋白的多个赖氨酸残基同时被单个泛素分子标记;而多聚泛素化修饰则是目标蛋白的单个赖氨酸残基被多个泛素分子标记(图1)。
泛素化修饰由E1泛素激活酶、E2泛素结合酶和E3泛素连接酶[3]的3步酶促反应协同作用实现。在哺乳动物中,目前已知其存在1个E1泛素激活酶、40个E2泛素结合酶和600多个E3泛素连接酶[4]。E1泛素激活酶通过游离泛素与ATP结合形成泛素-腺苷酸复合物,并将泛素转移至E2泛素结合酶。E2泛素结合酶接收由E1泛素激活酶传递的泛素,并与E3泛素连接酶协同作用完成对目标蛋白的泛素化修饰。在此过程中,E3泛素连接酶是决定目标蛋白是否进行泛素化修饰的主要因素。根据泛素的结构和转移机制的不同,E3泛素连接酶可分为3个主要家族:指环结构域(really interesting new gene domain,RING)、E6-AP羧基端同源结构域和RING之间的结构域 [5]。每个家族都有其独有的特征和功能,为细胞中的泛素化修饰增加了多样性和复杂性。
泛素化修饰通常涉及1个或多个重复的泛素单元,泛素可以以不同的方式连接形成不同类型的链状结构。其中,K48和K63位多聚泛素化修饰是研究最为广泛的类型[6]。K48位多聚泛素化修饰是通过连接泛素分子的K48位点来形成链状结构,促使发生泛素化修饰的蛋白质被标记上降解信号,进而被蛋白酶体降解。K63位多聚泛素化修饰通过连接泛素分子的K63位点来形成链状结构,涉及信号传导、DNA损伤修复和细胞内物质转运等生物学过程。
-
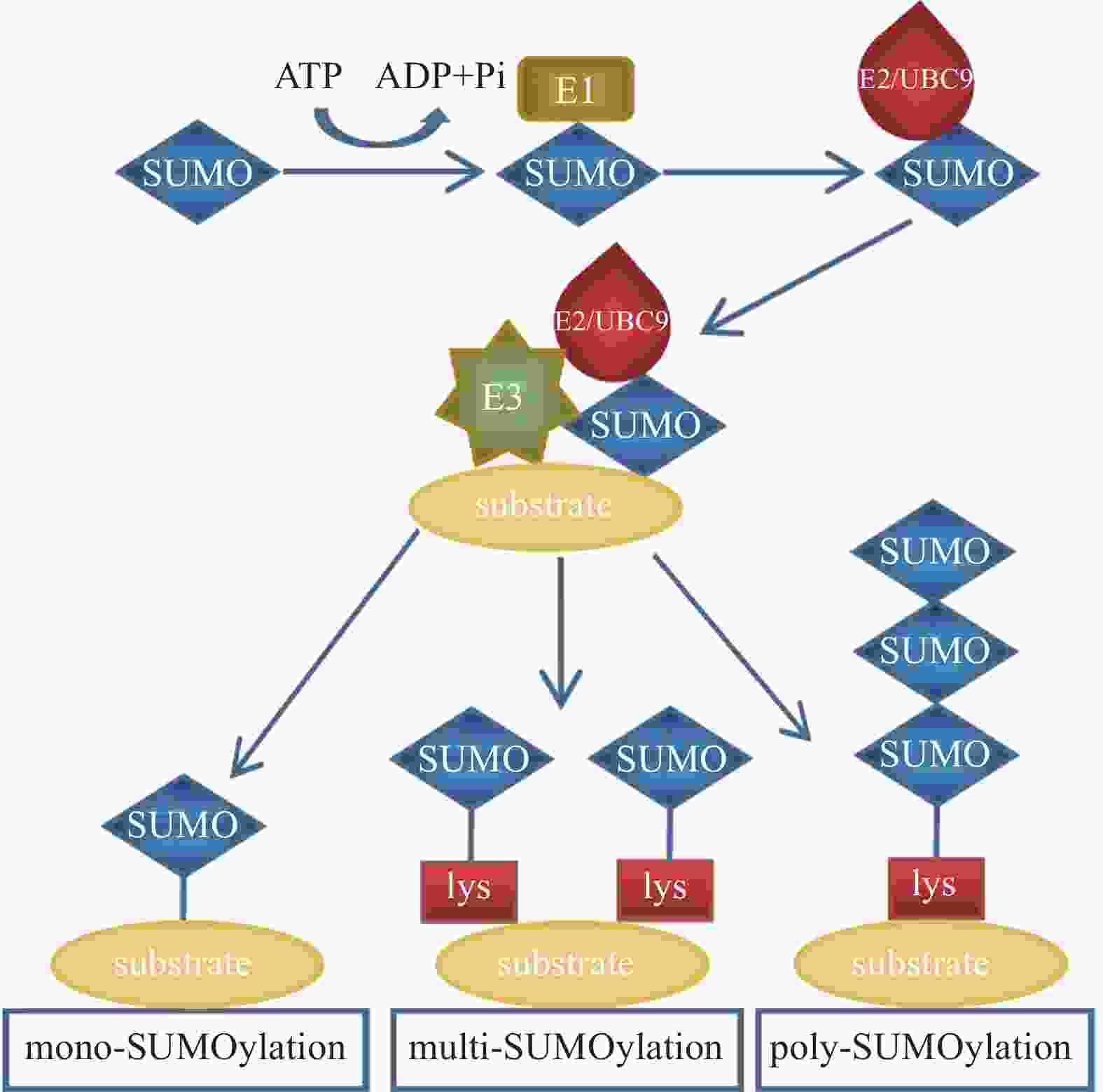
除泛素外,还存在许多结构和序列相似但功能不同的类泛素分子,SUMO是其中一类PTM因子,参与调控细胞中多种生物学过程[7]。SUMO家族包括SUMO1、SUMO2和SUMO3。其中SUMO1是最早被鉴定出的SUMO家族成员;SUMO2和SUMO3在结构上极为相似,仅有3个氨基酸不同,它们的同源性高达95%[8],因此常被写作SUMO2/3。未成熟的SUMO前体在偶联之前需要经过SUMO特异性蛋白酶1(SUMO-specific protease 1,SENP1)的切割,将其转化为成熟的SUMO蛋白[9]。SUMO与泛素的结合过程相似:首先由SUMO E1激活酶的2个亚基—SUMO活化酶亚基1(SAE1)和SUMO活化酶亚基2(SAE2)以ATP依赖的方式使成熟SUMO的C端二甘氨酸残基带电,活化的SUMO通过硫酯转移与SUMO E2结合酶UBC9相互作用; 之后SUMO E2结合酶UBC9通过SUMO E3连接酶直接将SUMO分子与底物蛋白的赖氨酸残基连接起来。其结合过程类似于泛素化修饰,SUMO化修饰也可以分为单SUMO化修饰、多SUMO化修饰和多聚SUMO化修饰(图2)。
SUMO以共价键形式连接到目标蛋白上,在影响目标蛋白结构和功能的同时,也能调节其亚细胞定位、相互作用等多种生物学特性。SUMO在细胞周期调控、DNA复制和修复、基因表达调节、细胞凋亡和神经元可塑性等生理过程中发挥重要作用[10]。同时其与内质网应激通路和炎症反应相关。SUMO在细胞信号传导和调控中具有重要作用,因此被认为是许多疾病的潜在治疗靶点,如心血管疾病[11]。目前,人们正在深入研究泛素化修饰和SUMO化修饰与疾病之间的关系,并寻求相关的治疗方法和策略,这使得泛素化修饰和SUMO化修饰在医学领域中具有广泛的研究价值和应用前景[12]。
-
IR导致DNA损伤,进而使细胞死亡或癌变[13]。DNA损伤类型主要包括DNA单链断裂、DNA双链断裂(DNA double-strand break,DSB)、DNA链间交联(DNA inter-strand crosslink,ICL)和DNA-蛋白质交联(DNA protein crosslinking,DPC)等。针对这些DNA损伤修复的方式主要有同源重组(homologous recombination,HR)修复、非同源末端连接(non-homologous end joining,NHEJ)修复、核苷酸切除修复(nucleotide excision repair,NER)、碱基切除修复(base excision repair,BER)和链间交联修复(inter-strand crosslink repair,ICLR)等。泛素化修饰和SUMO化修饰在这些损伤修复中发挥着重要作用。
-
DSB是最严重的DNA损伤类型之一[14],其主要有HR和NHEJ 2条修复途径。当IR导致DSB后,受损的DNA末端可以被减数分裂重组蛋白11(meiotic recombination protein11,Mre11)、辐射敏感蛋白50和Nijmegen断裂综合征蛋白1(Nijmegen breakage syndrome protein 1,Nbs1)形成的MRN复合物或Ku70/Ku80二聚体复合物识别[15],从而使HR或NHEJ途径得到修复。在HR修复过程中,关键步骤是DNA末端切除和同源互配。当MRN复合物被招募到损伤位点上时,Mre11通过核酸解旋反应、核酸内切酶活性等多种机制,实现了DNA的逐步降解,并促进后续的DNA末端加工和处理过程。核酸外切酶Ⅰ(exonuclease Ⅰ)通常参与切除受损的双链DNA,形成长的单链DNA。单链DNA片段侵入同源染色体中的相应区域,并与之配对形成一个DNA结合复合物,断裂的DNA链从同源染色体获取所需的遗传信息,进行DNA链的延伸,当DNA复制完成后,DNA链间的空间配对分子解离,新合成的DNA链与其相应的DNA末端连接,最终完成修复。当MRN复合物被招募到受损的DNA上后,在E3泛素连接酶Skp2的作用下,Nbs1发生K63位多聚泛素化修饰,进而招募共济失调毛细血管扩张症突变蛋白(ataxia-telangiectasia mutated protein,ATM)到损伤位点,ATM在第139位丝氨酸处磷酸化组蛋白H2AX,形成γ-H2AX[16]。随后,ATM激活后的复合物识别DNA损伤检查点介导因子1,从而启动DNA损伤应答(DNA damage response,DDR),并招募其他DDR因子,如受体相关蛋白80(receptor-associated protein 80,Rap 80)、p53结合蛋白1(53BP1)和乳腺癌易感基因1(breast cancer susceptibility gene 1,BRCA1)等。其中,在泛素相互作用基序(ubiquitin-interacting motif,UIM)的作用下,Rap80发生K63位多聚泛素化修饰,并与UIM相互作用[17],促进Rap80、ATM、Rad3相关蛋白1(ABR1)和BRCA1复合物的组装。BRCA1是一种功能多样的蛋白质,其含有一个RING结构域,可以和BARD1一起形成具有E3泛素连接酶活性的异源二聚体,并将泛素小分子连接到赖氨酸残基上。在DDR中,BRCA1-BARD1能够与多种底物发挥作用,如组蛋白H2A、H2AX和C端结合蛋白相互作用蛋白(CtIP)等[18],同时通过与SUMO E3连接酶PIAS1/4结合发生SUMO化修饰;与调节因子相互作用,影响染色质的结构、转录以及DNA修复的过程。
E3泛素连接酶在DDR过程中发挥重要的作用。当发生DSB损伤时,E3泛素连接酶RNF8被激活并转移到损伤位点,在组蛋白H2A或H2AX上发生K63位多聚泛素化修饰[19],形成初始的泛素信号;E3泛素连接酶RNF168进一步识别已泛素化修饰或SUMO化修饰的H2A蛋白,并在其组蛋白H2A上的K13~K15残基处进行泛素化修饰,从而放大泛素信号的强度[20];E3泛素连接酶RNF4和RFWD3与复制蛋白A(replication protein A,RPA)相互作用并促使其发生泛素化修饰,使RPA从DNA损伤位点去除,并抑制HR修复的发生。此外,前mRNA加工因子19(pre-mRNA processing factor 19,PRP19)也可以泛素化修饰RPA,并与共济失调毛细血管扩张突变基因Rad3相关激酶(ataxia telangiectasia and Rad3-related,ATR)相互作用蛋白(ATRIP)结合,激活ATR,促进HR修复。PRP19的过表达促进了RPA32和RPA70的泛素化修饰。在DDR过程中,RPA32以PRP19依赖的方式发生K63位多聚泛素化修饰[21]。RPA还可以发生SUMO化修饰,使RPA与Rad51结合,促进HR修复[22]。另外,泛素化修饰和SUMO化修饰可以与其他修饰(如磷酸化等)发生交叉调控,共同调节HR修复的效率和准确性[23]。
-
NHEJ修复是另一种常见的DSB修复机制,能迅速修复断裂的DNA双链结构。然而,由于缺乏同源模板的参与,常导致碱基序列改变[24]。Ku70和Ku80在NHEJ修复中形成Ku70/Ku80复合物,迅速结合到断裂的DNA末端上。SUMO化修饰可以增强Ku70/Ku80复合物的结合力,并激活DNA-依赖性蛋白激酶(DNA-dependent protein kinase,DNA-PK)的催化亚基,形成活性的DNA-PK复合物[25]。而E3泛素连接酶RNF144A能够对DNA-PK发生K48位多聚泛素化修饰和降解,从而促进DNA的损伤修复。DNA-PK招募DNA损伤修复因子到损伤部位,包括DNA末端突出端的核酸内切酶Artemis以及连接DNA平末端的DNA连接酶Ⅳ[26]。DNA-PK通过磷酸化作用募集DNA修复蛋白,如X射线修复交叉互补蛋白4(X-ray repair cross-complementing protein 4,XRCC4)和类XRCC4因子等,并与DNA连接酶Ⅳ形成复合物。此外,E3泛素连接酶TRIM28泛素化修饰XRCC4,并调控其稳定性和活性,提高修复断裂DNA的效率。同时,XRCC4通过与E3泛素连接酶Fbw7发生K63位多聚泛素化修饰以增强与Ku复合物的结合力,促进NHEJ的修复[27]。为了高效及时修复DSB,Ku80受到E3泛素连接酶RNF8或RNF138介导的K48位多聚泛素化修饰的调控,其可以将Ku70/Ku80复合物从DSB中迅速移除,而Ku70则受到SUMO E3连接酶PIAS1的调控,发生SUMO化修饰;在E3泛素连接酶RNF168的作用下,发生K63位多聚泛素化修饰并促进53BP1在损伤位点的招募,参与NHEJ的修复[28]。
-
当IR引起链间交联损伤时,细胞会启动ICLR机制。其主要依赖于范可尼贫血(Fanconi anaemia,FA)-BRCA通路以及核苷酸交换修复[29]。具体而言,当FA互补群M蛋白(Fanconi complementation group M protein,FANCM)结合到链间交联后,FANCM会招募其他相关蛋白质,如FA互补群蛋白Ⅰ(Fanconi anemia complementation group Ⅰ protein,FANCI)、FA互补群蛋白D2(Fanconi anemia complementation group D2 protein,FANCD2)和FA核心复合物相关蛋白20(Fanconi anemia core complex associated protein 20,FAAP20)等,形成核心的FA复合物[30]。其中,FANCI和FANCD2可以在E3泛素连接酶Rad18作用下与单个泛素连接,发生单泛素化修饰[31],促进FANCI和FANCD2的相互作用,从而促进FA复合物的组装和激活。另外,FANCD2在DSB修复过程中还能被单个SUMO分子连接,发生单SUMO化修饰。单SUMO化修饰促进FANCD2与其他FA蛋白形成复合物,从而调节DNA损伤修复通路的激活[32];FAAP20可以在E3泛素连接酶Fbw7作用下通过磷酸化依赖性方式发生泛素化修饰和降解,当FA修复完成时清除FAAP20。同时E3泛素连接酶Fbw7的活性受到糖原合成酶激酶3(GSK3)的调控,其可以磷酸化Fbw7,从而增加泛素连接酶Fbw7的活性,促进FAAP20的降解[33],协调ICLR。
-
IR也会导致胸腺嘧啶二聚体的形成,这种损伤可以通过2种不同的NER机制进行修复,即全基因组核苷酸切除修复(global genome nucleotide excision repair,GG-NER)和转录偶联核苷酸切除修复(transcription coupled nucleotide excision repair,TC-NER)[34],它们在识别和修复DNA损伤的过程中有所不同。GG-NER是一种常规的DNA修复机制,能够识别和修复DNA中的各种类型的损伤,而TC-NER主要用于基因转录相关的DNA损伤。在GG-NER中,直接识别损伤并招募复合物,进行DNA序列切割和去除。然后,DNA聚合酶和DNA连接酶填补缺失的DNA序列,完成修复过程。TC-NER的修复过程与GG-NER类似,但其修复过程主要发生在已转录的基因区域附近,以确保及时修复与转录相关的损伤。GG-NER和TC-NER共同协调工作,维护DNA的完整性和细胞正常功能。
C组着色性干皮病偶联因子(xeroderma pigmentosum complementation group C,XPC)是NER中的重要修复蛋白之一[35],其负责在受损的DNA位点上启动NER。XPC的招募受不同修饰方式的调节,其中泛素化修饰是关键的一种方式。泛素特异性蛋白酶(ubiquitin-specific protease,USP)家族成员USP7、USP11和USP24等可以去除XPC的泛素化修饰,提高XPC的稳定性和功能,并促进NER复合物的形成。USP7可以调节NER中XPC的功能,并去除其他NER参与因子的泛素化修饰,如DNA损伤结合蛋白2(DNA damage-binding protein 2,DDB2);USP11延长其在受损DNA上的存在时间,提高NER的效率。而USP24则通过稳定DDB2,促进XPC的泛素化及NER的过程[36]。
增殖细胞核抗原(proliferation cell nuclear antigen,PCNA)是一个重要的细胞周期蛋白,参与DNA复制和修复过程。在NER中,PCNA发生SUMO化修饰后,可以调控NER复合物的组装,并与DNA聚合酶δ相互作用。DNA聚合酶δ是NER中DNA链合成的主要酶,负责替换受损的DNA片段。通过与SUMO化修饰的PCNA相互作用,DNA聚合酶δ能够定位到损伤位点,促进NER。
-
当IR导致细胞中单个碱基损伤后,细胞会启动BER。在BER中,多个蛋白参与其中发挥作用,如PCNA发生泛素化修饰后能够促进其与其他修复因子的相互作用,从而提高DNA损伤修复效率,招募和稳定其他DNA修复酶,如DNA碱基切割内切酶1(apurinic/apyrimidinic endonuclease 1,APE1)[37]和多聚ADP核糖转移酶[poly(ADP-ribose)polymerase 1,PARP1][38]。APE1被泛素化修饰,通过泛素-蛋白酶体途径进行降解,这将影响其作为转录因子的活性,从而调节DNA修复和基因表达;当发生单个碱基损伤时,PARP1对损伤位点进行识别,其会被激活并与损伤部位结合。PARP1的活性依赖于烟酰胺腺嘌呤二核苷酸(nicotinamide adenine dinucleotide,NAD+)的供应。PARP1的激活会耗尽细胞内的NAD+,从而使细胞处于能量枯竭状态。为了恢复细胞内的NAD+水平,PARP1与APE1相互作用,激活APE1的切割活性,增强APE1在BER中的作用。此外,酵母中的DNA糖基化酶(DNA glycosylase,TDG)[39]也参与了BER,并修复受损的DNA。SUMO化修饰可调控TDG的活性和DNA结合能力,降低TDG对修复的作用。因此,SUMO化修饰可被视为一种负向调节机制,抑制TDG的DNA修复功能[40]。
-
泛素和SUMO能够以共价键形式连接到目标蛋白,分别发生泛素化修饰和SUMO化修饰,它们在HR修复、NHEJ修复、ICLR、NER和BER等DNA损伤修复中发挥着重要作用。例如泛素化修饰和SUMO化修饰能招募激酶到DNA损伤位点,进而启动DDR过程;招募其他结合蛋白参与DNA修复,促进DNA损伤修复复合物的组装;与调节因子相互作用,影响染色质结构和调控转录等。然而目前对DNA损伤修复机制的了解仍然不够全面,因此研究蛋白修饰对基因表达和DNA损伤修复的调节机制,将有助于个体化放射治疗策略的实施,根据患者的表观遗传特征进行治疗。未来的研究工作需要深入探究泛素化修饰和SUMO化修饰在DNA损伤修复过程中的精细调控机制,包括它们如何调节DNA修复蛋白的功能、相互作用等方面,为DNA损伤相关疾病提供更为精准而有效的治疗策略[41]。随着泛素化修饰和SUMO化修饰的进一步研究,更多泛素化修饰和SUMO化修饰原理及模型有待发现,这将有助于我们更好地理解它们在基因组稳定性维护和肿瘤治疗中的作用机制,并为相关疾病的预防和治疗提供新的思路和方法。
利益冲突 所有作者声明无利益冲突
作者贡献声明 张焕腾负责文献的查阅、综述的撰写;孙毓筱、徐畅负责综述的修改与审阅
泛素化修饰和SUMO化修饰在电离辐射诱导的DNA损伤修复中的作用机制
Mechanism of ubiquitination modification and SUMOylation modification in DNA damage repair induced by ionizing radiation
-
摘要: 泛素和小泛素样修饰物(SUMO)以共价键形式连接到特定的蛋白底物上,进行泛素化修饰或SUMO化修饰,通过调控蛋白质的稳定性、活性、定位或相互作用来影响多种细胞活动,其中包括DNA损伤修复、细胞周期、细胞凋亡和免疫应答等。当细胞受到DNA损伤时,泛素化修饰和SUMO化修饰能够调控相关蛋白质的功能和相互作用,参与到DNA损伤修复和信号传导的过程中。这些修饰作用对于维持基因组的完整性至关重要。近年来,研究发现泛素化修饰和SUMO化修饰在DNA损伤修复中都发挥着重要作用。笔者对这些作用机制进行综述,为深入了解电离辐射诱导的DNA损伤修复机制提供参考。
-
关键词:
- 泛素 /
- 小泛素相关修饰蛋白质类 /
- 辐射,电离 /
- DNA损伤 /
- DNA修复
Abstract: Ubiquitin and small ubiquitin-like modifier (SUMO) can be covalently attached to specific protein substrates, undergo ubiquitination modification and SUMOylation modification, and affecting their stability, activity, localization or interaction, thus regulating various cell activities, including DNA damage repair, cell cycle, apoptosis and immune responses. When cells experience DNA damage, ubiquitination modification and SUMOylation modification regulate the function and interaction of relevant proteins, thereby participating in the process of DNA damage repair and signal transduction. These modifications are indispensable for maintaining genome integrity. Recent studies have revealed that ubiquitination modification and SUMOylation modification in these repairs. The author reviews these roles, so as to provide a reference for in-depth understanding of the ionizing radiation-induced DNA damage repair mechanism.-
Key words:
- Ubiquitin /
- Small ubiquitin-related modifier proteins /
- Radiation, ionizing /
- DNA damage /
- DNA repair
-
[1] Pellegrino NE, Guven A, Gray K, et al. The next frontier: translational development of ubiquitination, SUMOylation, and NEDDylation in cancer [J/OL]. Int J Mol Sci, 2022, 23(7): 3480[2023-08-29]. https://www.mdpi.com/1422-0067/23/7/3480. DOI: 10.3390/ijms23073480. [2] Buneeva O, Medvedev A. Atypical ubiquitination and Parkinson's disease [J/OL]. Int J Mol Sci, 2022, 23(7): 3705[2023-08-29]. https://www.mdpi.com/1422-0067/23/7/3705. DOI: 10.3390/ijms23073705. [3] Toma-Fukai S, Shimizu T. Structural diversity of ubiquitin E3 ligase [J/OL]. Molecules, 2021, 26(21): 6682[2023-08-29]. https://www.mdpi.com/1420-3049/26/21/6682. DOI: 10.3390/molecules26216682. [4] Blaquiere N, Villemure E, Staben ST. Medicinal chemistry of inhibiting RING-type E3 ubiquitin ligases[J]. J Med Chem, 2020, 63(15): 7957−7985. DOI: 10.1021/acs.jmedchem.9b01451. [5] Licchesi JDF, Laman H, Ikeda F, et al. Editorial: E3 ubiquitin ligases: from structure to physiology [J/OL]. Front Physiol, 2020, 11: 621053[2023-08-29]. https://www.frontiersin.org/journals/physiology/articles/10.3389/fphys.2020.621053/full. DOI: 10.3389/fphys.2020.621053. [6] Tracz M, Bialek W. Beyond K48 and K63: non-canonical protein ubiquitination[J]. Cell Mol Biol Lett, 2021, 26(1): 1. DOI: 10.1186/s11658-020-00245-6. [7] Liu H, Craig SEL, Molchanov V, et al. SUMOylation in skeletal development, homeostasis, and disease[J/OL]. Cells, 2022, 11(17): 2710[2023-08-29]. https://www.mdpi.com/2073-4409/11/17/2710. DOI: 10.3390/cells11172710. [8] Tomanov K, Julian J, Ziba I, et al. SUMO conjugation and SUMO chain formation by plant enzymes[J]. Methods Mol Biol, 2023, 2581: 83−92. DOI: 10.1007/978-1-0716-2784-6_7. [9] Zhou W, Hu GL, He JL, et al. SENP1-Sirt3 signaling promotes α-ketoglutarate production during M2 macrophage polarization [J/OL]. Cell Rep, 2022, 39(2): 110660[2023-08-29]. https://www.sciencedirect.com/science/article/pii/S2211124722004120. DOI: 10.1016/j.celrep.2022.110660. [10] Liu J, Wu ZS, Han D, et al. Mesencephalic astrocyte-derived neurotrophic factor inhibits liver cancer through small ubiquitin-related modifier (SUMO)ylation-related suppression of NF-κB/snail signaling pathway and epithelial-mesenchymal transition[J]. Hepatology, 2020, 71(4): 1262−1278. DOI: 10.1002/hep.30917. [11] Xiao H, Zhou H, Zeng GF, et al. SUMOylation targeting mitophagy in cardiovascular diseases[J]. J Mol Med(Berl), 2022, 100(11): 1511−1538. DOI: 10.1007/s00109-022-02258-4. [12] Hotz P W, Müller S, Mendler L. SUMO-specific Isopeptidases tuning cardiac SUMOylation in health and disease [J/OL]. Front Mol Biosci, 2021, 8: 786136[2023-08-29]. https://www.frontiersin.org/articles/10.3389/fmolb.2021.786136/full. DOI: 10.3389/fmolb.2021.786136. [13] Yu H, Kondo Y, Fujii K, et al. Establishment of a method for investigating direct and indirect actions of Ionizing Radiation using scavenger-free plasmid DNA[J]. Radiat Res, 2022, 197(6): 594−604. DOI: 10.1667/rade-21-00057.1. [14] Park YJ, Kim TS, Kim EH, et al. Ribosomal protein S3 is a novel negative regulator of non-homologous end joining repair of DNA double-strand breaks[J]. FASEB J, 2020, 34(6): 8102−8113. DOI: 10.1096/fj.201903245R. [15] Menolfi D, Zha S. ATM, ATR and DNA-PKcs kinases-the lessons from the mouse models: inhibition ≠ deletion [J/OL]. Cell Biosci, 2020, 10: 8[2023-08-29]. https://cellandbioscience.biomedcentral.com/articles/10.1186/s13578-020-0376-x. DOI: 10.1186/s13578-020-0376-x. [16] Zhang CY, Guan YG, Zou JN, et al. Histone methyltransferase MLL1 drives renal tubular cell apoptosis by p53-dependent repression of E-cadherin during cisplatin-induced acute kidney injury [J/OL]. Cell Death Dis, 2022, 13(9): 770[2023-08-29]. https://www.nature.com/articles/s41419-022-05104-0. DOI: 10.1038/s41419-022-05104-0. [17] Liu YZ, Xu QS, Deng F, et al. HERC2 promotes inflammation-driven cancer stemness and immune evasion in hepatocellular carcinoma by activating STAT3 pathway[J]. J Exp Clin Cancer Res, 2023, 42(1): 38. DOI: 10.1186/s13046-023-02609-0. [18] Hu Q, Botuyan MV, Zhao DB, et al. Mechanisms of BRCA1-BARD1 nucleosome recognition and ubiquitylation[J]. Nature, 2021, 596(7872): 438−443. DOI: 10.1038/s41586-021-03716-8. [19] Burge RJ, Damianou A, Wilkinson AJ, et al. Leishmania differentiation requires ubiquitin conjugation mediated by a UBC2-UEV1 E2 complex[J]. PLoS Pathog, 2020, 16(10): e1008784. DOI: 10.1371/journal.ppat.1008784. [20] Kelliher JL, West KL, Gong QG, et al. Histone H2A variants alpha1-extension helix directs RNF168-mediated ubiquitination [J/OL]. Nat Commun, 2020, 11(1): 2462[2023-08-29]. https://www.nature.com/articles/s41467-020-16307-4. DOI: 10.1038/s41467-020-16307-4. [21] Hou PP, Yang KX, Jia PH, et al. A novel selective autophagy receptor, CCDC50, delivers K63 polyubiquitination-activated RIG-I/MDA5 for degradation during viral infection[J]. Cell Res, 2021, 31(1): 62−79. DOI: 10.1038/s41422-020-0362-1. [22] Zhu SH, Hou J, Gao HY, et al. SUMOylation of HNRNPA2B1 modulates RPA dynamics during unperturbed replication and genotoxic stress responses [J]. Mol Cell, 2023, 83(4): 539-555. e7. DOI:10.1016/j.molcel.2023.01.003. [23] Qin Y, Li Q, Liang WB, et al. TRIM28 SUMOylates and stabilizes NLRP3 to facilitate inflammasome activation [J/OL]. Nat Commun, 2021, 12(1): 4794[2023-08-29]. https://www.nature.com/articles/s41467-021-25033-4. DOI: 10.1038/s41467-021-25033-4. [24] Scully R, Panday A, Elango R, et al. DNA double-strand break repair-pathway choice in somatic mammalian cells[J]. Nat Rev Mol Cell Biol, 2019, 20(11): 698−714. DOI: 10.1038/s41580-019-0152-0. [25] Puustinen P, Keldsbo A, Corcelle-Termeau E, et al. DNA-dependent protein kinase regulates lysosomal AMP-dependent protein kinase activation and autophagy[J]. Autophagy, 2020, 16(10): 1871−1888. DOI: 10.1080/15548627.2019.1710430. [26] Watanabe G, Lieber MR, Williams DR. Structural analysis of the basal state of the Artemis: DNA-PKcs complex[J]. Nucleic Acids Res, 2022, 50(13): 7697−7720. DOI: 10.1093/nar/gkac564. [27] Guo GJ, Gao M, Gao XC, et al. Reciprocal regulation of RIG-I and XRCC4 connects DNA repair with RIG-I immune signaling [J/OL]. Nat Commun, 2021, 12(1): 2187[2023-08-29]. https://www.nature.com/articles/s41467-021-22484-7. DOI: 10.1038/s41467-021-22484-7. [28] Jiang QQ, Foglizzo M, Morozov YI, et al. Autologous K63 deubiquitylation within the BRCA1-A complex licenses DNA damage recognition[J]. J Cell Biol, 2022, 221(9): e202111050. DOI: 10.1083/jcb.202111050. [29] Fiesco-Roa MO, Giri N, Mcreynolds LJ, et al. Genotype-phenotype associations in Fanconi anemia: a literature review[J]. Blood Rev, 2019, 37: 100589. DOI: 10.1016/j.blre.2019.100589. [30] Zhang Y, Li J, Zhou Y, et al. And-1 coordinates with the FANCM complex to regulate Fanconi anemia signaling and cisplatin resistance[J]. Cancer Res, 2022, 82(18): 3249−3262. DOI: 10.1158/0008-5472.Can-22-0769. [31] Li LD, Tan W, Deans AJ. Structural insight into FANCI-FANCD2 monoubiquitination[J]. Essays Biochem, 2020, 64(5): 807−817. DOI: 10.1042/ebc20200001. [32] Germoglio M, Valenti A, Gallo I, et al. In vivo analysis of FANCD2 recruitment at meiotic DNA breaks in Caenorhabditis elegans [J/OL]. Sci Rep, 2020, 10(1): 103[2023-08-29]. https://www.nature.com/articles/s41598-019-57096-1. DOI: 10.1038/s41598-019-57096-1. [33] Wang JM, Chan B, Tong M, et al. Prolyl isomerization of FAAP20 catalyzed by PIN1 regulates the Fanconi anemia pathway[J]. PLoS Genet, 2019, 15(2): e1007983. DOI: 10.1371/journal.pgen.1007983. [34] Mullenders LHF. Solar UV damage to cellular DNA: from mechanisms to biological effects[J]. Photochem Photobiol Sci, 2018, 17(12): 1842−1852. DOI: 10.1039/c8pp00182k. [35] Mu H, Geacintov NE, Broyde S, et al. Molecular basis for damage recognition and verification by XPC-RAD23B and TFIIH in nucleotide excision repair[J]. DNA Repair (Amst), 2018, 71: 33−42. DOI: 10.1016/j.dnarep.2018.08.005. [36] Zhu QZ, Ding N, Wei SC, et al. USP7-mediated deubiquitination differentially regulates CSB but not UVSSA upon UV radiation-induced DNA damage[J]. Cell Cycle, 2020, 19(1): 124−141. DOI: 10.1080/15384101.2019.1695996. [37] Zhang Q, Yang LJ, Gao H, et al. APE1 promotes non-homologous end joining by initiating DNA double-strand break formation and decreasing ubiquitination of artemis following oxidative genotoxic stress [J/OL]. J Transl Med, 2023, 21(1): 183[2023-08-29]. https://translational-medicine.biomedcentral.com/articles/10.1186/s12967-023-04022-9. DOI: 10.1186/s12967-023-04022-9. [38] Alemasova EE, Lavrik OI. Poly(ADP-ribosyl)ation by: reaction mechanism and regulatory proteins[J]. Nucleic Acids Res, 2019, 47(8): 3811−3827. DOI: 10.1093/nar/gkz120. [39] Steinacher R, Barekati Z, Botev P, et al. SUMOylation coordinates BERosome assembly in active DNA demethylation during cell differentiation[J]. Embo J, 2019, 38(1): e99242. DOI: 10.15252/embj.201899242. [40] Wang DP, Wu W, Callen E, et al. Active DNA demethylation promotes cell fate specification and the DNA damage response[J]. Science, 2022, 378(6623): 983−989. DOI: 10.1126/science.add9838. [41] Park J, Cho J, Song EJ. Ubiquitin-proteasome system (UPS) as a target for anticancer treatment[J]. Arch Pharm Res, 2020, 43(11): 1144−1161. DOI: 10.1007/s12272-020-01281-8. -





 下载:
下载: